Accueil
Sommaire
Le Master
Le Master de Bioinformatique est une formation en deux ans, proposant à partir de la rentrée 2022, deux parcours :
- Le parcours Bioinformatique et biologie des Systèmes (BBS)
- Le parcours Bioinformatique et Génomique Environnementale (BGE). Ce parcours est mutualisé avec le master mention Biologie, Ecologie et Evolution.
La plaquette du master fournit un descriptif résumé de la formation.
Le recrutement des étudiants se fait dès le M1. Les mentions de Licence conseillées sont :
- Pour le parcours BBS :
- Licence Sciences de la Vie
- Licence d'Informatique
Les candidatures d’étudiant.e titulaire d’une licence montrant une forte interdisciplinarité Sciences de la vie, Mathématique et Informatique, ou titulaire d’une double licence Biologie-Informatique ou d’une licence Sciences de la vie ayant suivi le parcours BIOMIP de l’UT3 sont particulièrement adaptées à la poursuite d’étude dans le parcours BBS.
- Pour le parcours BGE :
- Licence Sciences de la Vie
Les candidatures d’étudiant.e titulaire d’une licence Sciences de la vie ayant suivi les parcours Biodiversité et Biologie Environnementale ou Biologie des populations/organismes (ou équivalent) d’UT3 sont particulièrement adaptées à la poursuite d’étude dans le parcours BGE .
Admission et candidature
Depuis la rentrée de septembre 2017, une nouvelle procédure d'admission a été mise en place au niveau national. le recrutement s’effectue à l’entrée de la première année du Master (M1).
- Les étudiants font le choix de leur Master dès la Licence.
- Des capacités d’accueil sont mises en place pour chacune des mentions et des parcours de masters.
- L’admission en 1re année de Master est subordonnée à l’examen d’un dossier du candidat.
La page principale du site de l'université pour les candidatures en master est http://www.univ-tlse3.fr/inscriptions/candidature-en-licence-ou-master-623443.kjsp?RH=1510909053704
Candidature en Master 1
Pour les titulaires d'un diplôme français, il faut déposer un dossier de candidature sur la plateforme MonMaster : https://www.monmaster.gouv.fr
Le dossier de candidature est constitué des pièces suivantes :
- un dossier détaillé du cursus suivi par le candidat permettant d’apprécier les objectifs et compétences visées par la formation antérieure ;
- les diplômes, certificats, relevés de notes permettant d’apprécier la nature et le niveau des études suivies ;
- une lettre de motivation exposant le projet professionnel et justifiant son adéquation avec la formation envisagée.
Pour les titulaires d'un diplôme étranger, les informations sont sur la page https://www.univ-tlse3.fr/candidature-en-m1-pour-les-titulaires-d-un-diplome-obtenu-a-l-etranger-1
Attention, si vous devez déposer votre candidature sur eCandidat bien regarder le liste des pièces à fournir car des informations complémentaires sur votre formation à l'étranger vous sont demandées.
Prérequis en connaissances et compétences
Pour le parcours Bioinformatique et Biologie des Systèmes
les prérequis en connaissances et compétences requises pour réussir dans cette formation sont :
- Maîtriser les concepts théoriques en génétique, génomique et biologie moléculaire
L’étudiant.e issu.e d’une licence de Sciences de la Vie doit maîtriser ces concepts pour pouvoir réaliser une interprétation et une synthèse rationnelle des données issues d’approche globale à haut débit.
- Être sensibilisé au traitement informatique des données biologiques
Idéalement, l’étudiant.e issu.e d’une licence de Sciences de la Vie devrait avoir eu une première expérience en bioanalyse et/ou bioinformatique l’ayant confronté au traitement informatique des données biologiques.
- Disposer d’une base solide sur les fondamentaux en informatique
L’étudiant.e issu.e d’une licence d’informatique devra maitriser l’algorithmique, la, les bases de données et posséder de bonnes notions sur les méthodes d’apprentissage automatique.
- Disposer d’une culture générale en Biologie
L’étudiant.e issu.e d’une licence d’Informatique devra posséder des connaissances de base en Biologie, de préférence moléculaire montrant leur sensibilisation à cette discipline.
- Disposer d’un intérêt fort pour les mathématiques/statistiques
La biologie des systèmes utilise fortement les mathématiques pour l’intégration de grands jeux de données hétérogènes et l’étude du comportement dynamique de réseaux (conception de modèles mathématiques explicatifs et prédictifs).
- Faire preuve d’interdisciplinarité
L’étudiant.e, à terme, devra concevoir des réponses méthodologiques informatiques et/ou mathématiques adaptées à la résolution de questions biologiques liées à l'analyse de données complexes. L’interdisciplinarité sera donc présente tout au long du cursus.
- Faire preuve de motivation
L’étudiant.e devra expliciter la cohérence et l’adéquation de son projet professionnel avec la formation proposée et démontrer sa capacité à s’investir.
- Faire preuve d’autonomie
La formation comportant la réalisation de nombreux projets individuels et en groupe, l’étudiant.e doit savoir travailler en autonomie avec une bonne gestion de son temps. De plus, elle ou il doit savoir interagir positivement lors du travail en équipe.
Pour le parcours Bioinformatique et Génomique Environnementale
les prérequis en connaissances et compétences requises pour réussir dans cette formation sont :
- Maîtriser les concepts-clés en biologie évolutive, écologie et génétique, et disposer de connaissances en génomique ou biologie moléculaire
L’étudiant.e doit maîtriser ces concepts pour tester des hypothèses et interpréter les données issues d’approches à haut débit en génomique environnementale.
- Être sensibilisé au traitement informatique des données biologiques / environnementales
Idéalement, l’étudiant.e devrait avoir eu une première expérience l’ayant confronté au traitement informatique des données biologiques. Il/Elle utilisera continuellement des outils logiciels et informatiques.
- Disposer d’un intérêt fort pour les biostatistiques/mathématiques
La génomique environnementale utilise fortement les biostatistiques pour la description, l’inférence et la prédiction à partir de grands jeux de données intraspécifiques et interspécifiques.
- Faire preuve d’interdisciplinarité
L’étudiant.e, à terme, devra utiliser ou concevoir des réponses méthodologiques adaptées à la résolution de questions écologiques et évolutives liées à l'analyse de données « omiques ». L’interdisciplinarité sera donc présente tout au long du cursus.
- Faire preuve de motivation
L’étudiant.e devra expliciter la cohérence et l’adéquation de son projet professionnel avec la formation proposée et démontrer sa capacité à s’investir.
- Faire preuve d’autonomie
La formation comportant la réalisation de nombreux projets individuels et en groupes, l’étudiant.e doit savoir travailler en autonomie avec une bonne gestion de son temps. De plus, elle ou il doit savoir interagir positivement lors du travail en équipe.
Candidature en Master 2
Cette année, seul le M2 du parcours Bioinformatique et Biologie des Systèmes ouvrira. En effet, le parcours Bioinformatique et Génomique Environnementale est un nouveau parcours proposé pour la première fois en M1 à la rentrée 2022.
Les dossiers sont à déposer sur la plateforme eCandidat entre le 2 mai et le 3 juin 2022 : https://ecandidat2.univ-tlse3.fr
Attention, seules les candidatures démontrant que les compétences et connaissances acquises dans la formation antérieure recouvrent en partie celles attendues à la fin du M1 de Bioinformatique de Toulouse seront recevables.
Pour les titulaires d'un diplôme étranger, les informations sont sur la page https://www.univ-tlse3.fr/candidature-en-m2-pour-les-titulaires-d-un-diplome-obtenu-a-l-etranger-1
Objectifs
L’objectif de ce master est de former des étudiant.e.s qui deviendront des scientifiques (chercheur.se.s ou ingénieur.e.s) capables de répondre aux questions de plus en plus complexes soulevées par les approches globales en biologie et faire face aux défis, aussi bien scientifiques que techniques, ainsi engendrés. Ceci nécessite donc d'acquérir des compétences multidisciplinaires, biologie, informatique et mathématiques, nécessaires pour œuvrer dans le domaine de la bioinformatique mais aussi dans ceux plus récents de la biologie des systèmes et de la génomique environnementale. Nous avons comme but de permettre aux étudiant.e.s d'acquérir :
- les connaissances et compétences en programmation et gestion des données pour accompagner des projets en biologie,
- les compétences en traitements mathématiques de grands jeux de données pour en extraire les informations pertinentes
- les démarches pour dégager, à partir de différentes sources de données hétérogènes, les relations entre objets dans le but d'inférer des réseaux biologiques
- des compétences pratiques par la réalisation de nombreux projets individuels et collectifs en plus d'un socle solide de connaissances théoriques.
L'apprentissage de la communication et de la transmission de leurs connaissances ainsi que l'exercice de l'esprit critique seront développés tout au long du cursus. A l'issue de cette formation, les étudiant.e.s auront acquis l’autonomie nécessaire pour conceptualiser les problèmes liés à l'analyse des données biologiques et pour mettre en place et/ou développer les réponses méthodologiques adaptées pour résoudre la question biologique posée.
L'évolution rapide des technologies dans le domaine des sciences de la vie et la généralisation de ces approches globales dans l'analyse du vivant génèrent dans les laboratoires privés et publics une demande accrue de jeunes cadres ou chercheur.se.s possédant une vision intégrée s'appuyant sur des connaissances et des compétences de plusieurs champs disciplinaires.
Parcours
Plus d'information sur le déroulement et l'organisation de la formation et des parcours sont disponibles sous l'onglet Formation
Débouchés et insertion professionnelle
Les détails sont fournis sous l'onglet insertion professionnelle et les débouchés
La bioinformatique
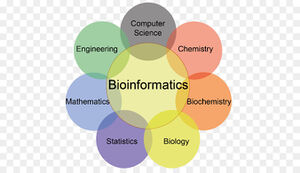
La bioinformatique est issue de la rencontre de la biologie, des mathématiques et de l'informatique et constitue une nouvelle branche de la biologie, appelée approche in silico, venant compléter les approches in situ, in vivo et in vitro. Discipline reconnue depuis les années 1990, elle avait cependant fait son apparition dès 1965 dans le cadre de la phylogénie moléculaire et s'est ensuite développée dans le cadre de l'analyse des séquences. En constante évolution, elle est aujourd’hui au cœur des approches de génomiques et post-génomiques.
Les programmes de séquençage systématique de génomes complets conjointement au développement d'approches globales à haut débit (transcriptomique, protéomique,...) ont conduit à la production de grandes masses de données mais aussi à un changement de cadre conceptuel où l'on passe d'une approche analytique à une approche synthétique : la fonction d'une entité biologique résulte de ces relations avec les autres acteurs du système. De fait, devant l'explosion de la production de données ainsi que celle de leur hétérogénéité, l'utilisation de l'informatique, dans le monde notamment de la génétique et de la biologie moléculaire, est devenue incontournable. En effet, les méthodes informatiques, souvent basées sur l'outil statistique, interviennent à de nombreux niveaux, de l'acquisition à l'exploitation des données en passant par le stockage et la diffusion de l'information. Ces différents niveaux forment des domaines bien identifiables mais toutefois reliés entre eux et alliés à la démarche expérimentale, ils ont pour but final et ambitieux de comprendre le vivant.
La bioinformatique a pour but d'étudier et d'interpréter les informations issues de l'étude du vivant et de permettre une synthèse rationnelle des données disponibles dans le double objectif d'énoncer des hypothèses généralisatrices et de formuler des prédictions à partir d'une approche par modélisation. Loin de n'être qu'une simple utilisation d'outils informatiques appliqués aux différents domaines de la biologie, l'originalité et la force de la bioinformatique s'exprime plus particulièrement dans le développement de nouvelles méthodes d'analyse des données en biologie ainsi que dans l'acquisition de nouvelles connaissances et l'émergence de nouveaux concepts. Sa maîtrise requiert une multiple compétence Biologie-Informatique-Mathématiques-Statistiques.
La biologie des systèmes
La biologie des systèmes est une voie de recherche récente s’intéressant aux interactions complexes rencontrés dans les systèmes biologiques et à la manière dont ces interactions donnent naissance à la fonction du système et sont impliquées dans son comportement.
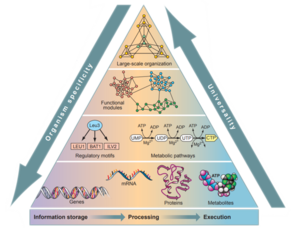
En effet, une fonction biologique ne résulte pas de la simple addition des propriétés de ses composants élémentaires (ARN, protéines, ...) mais des propriétés induites par leurs nombreuses interactions. Ceci nécessite donc la détermination et l’analyse de réseaux d’interactions complexes afin de les modéliser mathématiquement pour pouvoir étudier leur dynamique.
Le but ultime de cette approche est d’obtenir une compréhension holistique des phénomènes biologiques. De nombreuses étapes restent cependant à franchir. Il faut dans un premier temps identifier les composants pertinents du système, inférer le réseau de relations et réaliser ensuite une modélisation explicative et prédictive. Grâce aux progrès technologiques et l’avènement des approches à haut débit en biologie, les données peuvent être facilement produites. En effet, le séquençage des génomes permet de connaître l’organisation de ceux-ci, les approches, entre autres, du transcriptome (présence et abondance relative des ARN), du protéome (présence et abondance relative des protéines), du métabolome (ensemble des métabolites d’une cellule) permettent de dresser un bilan quantitatif et qualitatif des biomolécules dans des conditions expérimentales déterminées. On peut ainsi analyser l’expression du génome au cours du temps (lors du développement de la plante par exemple) ou dans des conditions environnementales différentes (par exemple, différentes conditions de stress). Quantité et hétérogénéité caractérisent les données issues de la génomique et de la post-génomique (appelée aussi génomique fonctionnelle).
Un premier enjeu central dans la biologie des systèmes concerne donc l’intégration de ces données, à savoir l’analyse et l’association de différents types de données et leur interprétation en termes biologiques. Par exemples, l’inférence de réseaux de gènes se ferra en associant les données d’analyse du génome (génomique) et les données du transcriptome, l’inférence de réseaux de régulation pourra être obtenue par intégration des données du transcriptome avec celles se rapportant aux interactions protéines-ADN, etc. L’exploitation et l’intégration efficace des données de génomique et de post-génomique reposent sur la bioinformatique. Pour étudier ensuite le comportement de ces réseaux, il faudra proposer des modèles mathématiques explicatifs et prédictifs qui seront confrontés aux résultats expérimentaux.
La biologie des systèmes se présente donc comme une démarche itérative et intégrative en combinant des approches expérimentales et théoriques. Dans un premier temps, les connaissances scientifiques disponibles (données produites par un ou plusieurs niveaux hiérarchiques de l'organisation des systèmes biologiques) seront intégrées dans un modèle mathématique préliminaire afin d’établir des prédictions sur le fonctionnement de ce système biologique dans de nouvelles conditions. En effet, des simulations numériques permettront d’étudier le comportement du système sous l’effet de différentes variations. Dans un second temps, certains éléments du système seront perturbés expérimentalement et les effets de cette perturbation analysés et confrontés aux résultats de la simulation numérique. Des contradictions entre résultats expérimentaux et théoriques conduiront à revisiter le modèle mathématique pour prendre en compte ces nouvelles connaissances. Le nouveau modèle sera testé de la même façon et le cycle sera répété jusque ce que les résultats observés et prédits se superposent. La modélisation doit être vue comme une aide à la compréhension du système et non comme une finalité en soi, c’est pourquoi les modèles doivent être explicatifs et prédictifs.
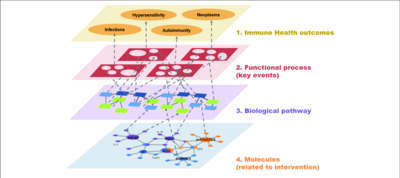
La santé est un champ d'application de la biologie des systèmes avec comme exemple de discipline associée la biologie des systèmes du cancer. Elle se distingue par la caractérisation moléculaires des tumeurs (tumorigenèse et le traitement des cancers). En effet, les mutations de l'ADN et l'expression des gènes participent à la progression tumorale, elles sont importantes pour mieux comprendre la biologie du cancer ainsi que les choix thérapeutiques envisagés. L'étude du système tumoral à différentes échelles (ADN, ARN, protéines, réseaux, cellules, organes, individu, population, ...) et de manière dynamique, propose un modèle qui s'appuie sur des interactions connues entre les composants du système (source Wikipedia). La figure ci-contre, montre une approche de biologie des systèmes dans le cadre de la santé immunitaire.
La Génomique Environnementale
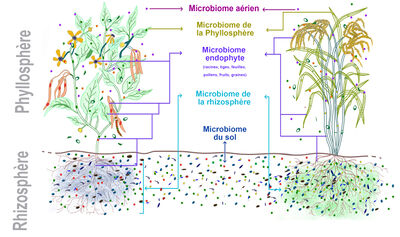
Les nouvelles technologies de séquençage haut-débit de l’ADN et de l’ARN sont désormais applicables à tout échantillon prélevé dans un environnement (sol, eau, air...) ou un compartiment biologique donné (rhizosphère, système digestif, fèces, restes organiques contemporains ou anciens, …), sans un nécessaire isolement préalable des organismes cibles composant l’échantillon. L’approche de génomique environnementale, générant une production massive de données relatives à la diversité taxonomique et fonctionnelle à différentes échelles spatiales et temporelles, modifie en profondeur les stratégies de recherche sur la biodiversité, l’écologie et l’évolution des organismes ainsi que sur la structure, la dynamique et le fonctionnement des écosystèmes. Ceci s’accompagne de défis à relever pour la bioinformatique : développement d’outils de gestion et de traitement des données afin d’établir une description précise et robuste de l’information biologique, développement de méthodes d’analyse de ces données, permettant de tester des hypothèses en biologie, écologie et évolution, et par extension de modéliser les systèmes biologiques dans un contexte environnement-dépendant.