Formation : Différence entre versions
m (→Master 2) |
m (→Le parcours Bioinformatique et Biologie des Systèmes) |
||
| (92 révisions intermédiaires par 2 utilisateurs non affichées) | |||
| Ligne 1 : | Ligne 1 : | ||
| − | [[Media:Plaquette.Master.Bioinformatique.2016-2021.pdf|Plaquette du Master]] | + | <!-- [[Media:Plaquette.Master.Bioinformatique.2016-2021.pdf|Plaquette du Master]] --> |
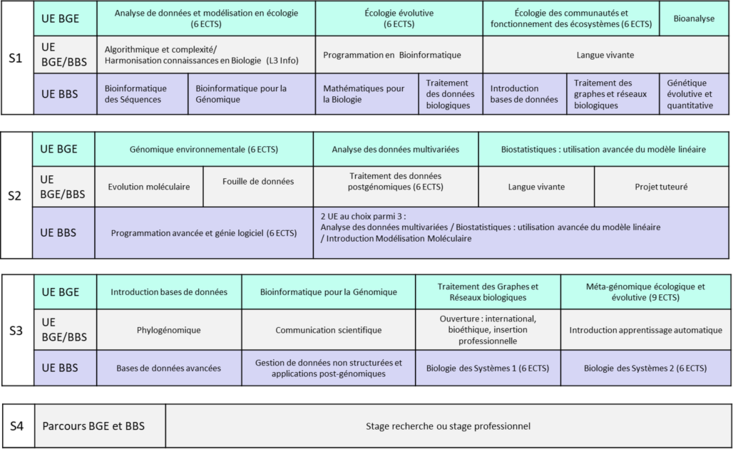
= Schéma général des études = | = Schéma général des études = | ||
| − | + | Le master de Bioinformatique se transforme à la rentrée 2022 et s'enrichit d'un second parcours, le parcours de '''Bioinformatique et Génomique Environementale''' en complément de celui déjà existant, le parcours de '''Bioinformatique et Biologie des Systèmes'''. | |
| − | + | [[Media:Plaquette_Master_Bioinformatique.pdf|La plaquette du master]] fournit un résumé descriptif de cette nouvelle offre de formation | |
| − | == Master 1 == | + | Cette section est donc en cours de construction et s'enrichira au cours du temps. |
| + | |||
| + | '''Le schéma ci-dessous présente l’organisation en UE de la mention par année et parcours''' : | ||
| + | |||
| + | * En vert les UE spécifiques au parcours « Bioinformatique et Génomique Environnementale » (BGE) | ||
| + | * En mauve les UE spécifiques au parcours « Bioinformatique et Biologie des Systèmes » (BBS) | ||
| + | * En gris les UE mutualisées entre les deux parcours | ||
| + | |||
| + | [[Fichier:Schema_mention.png|733px]] | ||
| + | |||
| + | |||
| + | == Syllabus== | ||
| + | |||
| + | Les syllabus ne sont pas encore disponibles sur le site de l'Université Paul Sabatier. En attendant leurs mises en ligne, merci de vous référer à la section Description des Unités d'Enseignements ci-dessous. | ||
| + | |||
| + | <!--* [[Media:Syllabus_M1_BioInfo.pdf|Syllabus de la première année de master]] ''' | ||
| + | * [[Media:Syllabus_M2_BioInfo.pdf|Syllabus de la deuxième année de master]] '''--> | ||
| + | |||
| + | == Le parcours Bioinformatique et Biologie des Systèmes == | ||
| + | |||
| + | === Objectifs de formation === | ||
| + | |||
| + | Le parcours BBS permettra aux étudiants d’acquérir (1) les connaissances en programmation et gestion des données pour accompagner des projets en biologie, (2) les compétences en traitements mathématiques de grands jeux de données pour en extraire les informations pertinentes, (3) les démarches pour dégager, à partir de différentes sources de données hétérogènes, les relations entre objets dans le but d'inférer des réseaux biologiques, (4) les méthodes de modélisation dynamique des réseaux biologiques pour analyser in silico leur comportement. | ||
| + | |||
| + | === Contenu des enseignements === | ||
| + | |||
| + | Ce parcours proposera une solide formation disciplinaire en Bioinformatique et en Biologie des Systèmes. Il mettra l'accent sur le traitement et l’intégration des différents types de données Omics, et sur l’inférence et la modélisation dynamique des réseaux de gènes afin de mieux comprendre le comportement dynamique du système biologique dans sa globalité. | ||
| + | |||
| + | Les principaux axes d’enseignement seront : | ||
| + | * en Informatique : Algorithmique et complexité, Programmation en Bioinformatique et Programmation avancée et Génie Logiciel, Introduction en bases de données et bases de données avancées,Gestion de données non structurées et les applications post-génomiques, Introduction à l'apprentissage automatique | ||
| + | * en Mathématiques : Mathématiques pour la Biologie, Traitement des données Biologiques, Analyses des données multivariées, Biostatistiques : utilisation avancée du modèle linéaire | ||
| + | * en Bioinformatique : Bioinformatique des séquences, Bioinformatique pour la Génomique, Traitement des graphes et réseaux, Traitement des données post-génomiques, Fouille de données | ||
| + | * en Evolution : évolution moléculaire, phylogénomique, Génétique évolutive et quantitative | ||
| + | * en Biologie des Systèmes | ||
| + | |||
| + | A la place de l'UE d'algorithmique et complexité, une UE Harmonisation des connaissances en Biologie sera proposée aux étudiant(e)s venant d'une licence d'Informatique. | ||
| + | |||
| + | La pédagogie fait appel le plus largement possible au travail en autonomie et/ou en groupe et s'appuie sur l'analyse des processus d'acquisition des connaissances scientifiques, des ateliers pratiques et la réalisation de projets. Le second semestre est consacré à un stage long (6 à 5 mois) dans des laboratoires de recherche ou en entreprise. | ||
| + | |||
| + | == Le parcours Bioinformatique et Génomique Environnementale == | ||
| + | Ce parcours, mutualisé entre le master de Bioinformatique et le master Biologie, Écologie et Évolution (BEE), est donc également proposé par le master BEE. | ||
| + | |||
| + | === Objectifs de formation === | ||
| + | |||
| + | Le parcours BGE permettra aux étudiants d’acquérir des bases conceptuelles et pratiques solides qui leur permettront de (i) traiter des données génomiques issues de prélèvements environnementaux, (ii) décrire la diversité taxonomique et fonctionnelle de ces prélèvements, (iii) modéliser les interactions entre les différentes composantes des systèmes biologiques décrits, ainsi qu’inférer leur évolution et prédire leur capacité d’adaptation, à l’aide de méthodes statistiques et d’apprentissage automatique. | ||
| + | |||
| + | === Contenu des enseignements === | ||
| + | |||
| + | Ce parcours proposera une solide formation disciplinaire en Bioinformatique, Biodiversité, Ecologie et Evolution, pour permettre de générer des connaissances sur le fonctionnement et l’évolution des organismes et écosystèmes, par l’analyse de l’information génétique ou génomique issue du séquençage non ciblé, dans un environnement donné. | ||
| + | |||
| + | Ainsi les principaux axes d’enseignement porteront sur : | ||
| + | * La génomique environnementale (ADN environnemental, ADN ancien, métabarcoding, métagénomique, génomique des populations, génomique écologique, génomique comparative) | ||
| + | * L’algorithmique et la programmation en bioinformatique, le traitement des données génomiques et post-génomiques, la phylogénomique, le traitement des graphes et réseaux. | ||
| + | * Les concepts clés en écologie évolutive, écologie microbienne et écologie des communautés et sur le fonctionnement des écosystèmes. | ||
| + | * Les traitements et modélisation statistiques des données en écologie et les modélisations par apprentissage automatique. | ||
| + | |||
| + | La pédagogie fait appel le plus largement possible au travail en autonomie et/ou en groupe et s'appuie sur l'analyse des processus d'acquisition des connaissances scientifiques, des ateliers pratiques et la réalisation de projets. Le second semestre est consacré à un stage long (6 à 5 mois) dans des laboratoires de recherche ou en entreprise. | ||
| + | |||
| + | == Description des Unités d'Enseignement== | ||
| + | <font face = "Times New Roman" size = "4"> | ||
| + | Le parcours et l'année sont indiquées pour chaque UE<br> | ||
| + | === UE Master 1 === | ||
| + | </font> | ||
| + | ====Algorithmique et complexité==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BBS et BGE''' <br> | ||
| + | '''ECTS : 3'''<br> | ||
| + | '''Objectifs'''<br> | ||
| + | Cette UE est pour les étudiants provenant de biologie. Les autres étudiants suivront l'UE d'Harmonisation des connaissances en biologie. | ||
| + | Décomposer un programme en éléments de plus petite taille. | ||
| + | Analyser le comportement de programmes simples fondés sur les variables, expressions, affectations, E/S, structure de contrôle, fonctions... | ||
| + | Créer des algorithmes résolvant des problèmes simples : transcription ADN en ARN, traduction en protéines. | ||
| + | Implémenter des algorithmes, les tester et les déboguer. | ||
| + | Comprendre les notions et les techniques qui permettent d’analyser la complexité d’algorithmes. | ||
| + | Initiation aux systèmes Unix : gestion des fichiers, commandes de base. <br> | ||
| + | '''Prérequis'''<br> | ||
| + | Mathématiques élémentaires<br/> | ||
| + | '''Contenu'''<br> | ||
| + | ''Algorithmique''<br> | ||
| + | * Syntaxe élémentaire, notion de variables, constantes... | ||
| + | * Expressions et affectations | ||
| + | * Entrées-sorties simples | ||
| + | * Structures de contrôle | ||
| + | * Types de données (entiers, réels, chaîne de caractères, tableaux, listes, dictionnaires) | ||
| + | * Fonctions | ||
| + | ''Concepts de la programmation''<br> | ||
| + | * Concept d'algorithme | ||
| + | * Types d'erreur | ||
| + | * Compréhension des programmes | ||
| + | * Algorithmes simples (moyenne, min, max d’une liste, manipulation de séquences ADN, transcription ARN, traduction protéines) | ||
| + | * Stratégies de résolution de problèmes | ||
| + | ''Complexité''<br> | ||
| + | Introduction des notions et techniques qui permettent d’analyser la complexité d’algorithmes. | ||
| + | Complexité de boucles, ''pour'' ou ''tantque'', dans le cas le pire et le cas moyen. | ||
| + | ''Systèmes Unix'' <br> | ||
| + | * introduction aux système de gestion de fichiers | ||
| + | * commandes SHELL de base, notions de filtrages et d'entrées sorties | ||
| + | '''Références'''<br> | ||
| + | Algorithmes - Notions de base. Thomas H. Cormen. Dunod, 2013.<br> | ||
| + | Unix, Initiation et utilisation : Colin, Armspach, Ostré-Waerzeggers, Dunod, 2004 <br> | ||
| + | <!--competences=Modéliser un problème simple et savoir implémenter un algorithme pour le résoudre. | ||
| + | Analyser la complexité d’algorithmes itératifs et de structures de données.| | ||
| + | keywords=Algorithmique ; structures ; instructions conditionnelles ; séquences nucléiques et protéiques ; annotations structurées ; complexité asymptotique | ||
| + | --> | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Programmation en bioinformatique==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BBS et BGE''' <br> | ||
| + | '''ECTS : 3'''<br> | ||
| + | '''Objectifs'''<br> | ||
| + | Apprentissage de la programmation structurée avec des langages impératifs. Savoir utiliser les structures de contrôle (tests, boucles), les types élémentaires et les structures de données classiques (listes, dictionnaires, etc). Savoir structurer son code en fonctions et transformer les scripts en modules réutilisables. Mise en œuvre en utilisant un langage impératif (python). Utilisation de modules/paquets/bibliothèques de programmation dédiés à la bioinformatique, notamment pour la manipulation de séquences (nucléiques et protéiques) en utilisant les formats standards en bioinformatique (FASTA, GenBank, etc), ainsi que l’exploitation de diverses sources et formats de données. Introduction à la programmation objet. Savoir utiliser une interface de programmation.<br> | ||
| + | '''Prérequis''' <br> | ||
| + | UE Algorithmique et complexité <br> | ||
| + | '''Contenu'''<br> | ||
| + | Notions fondamentales | ||
| + | |||
| + | * Expressions : types de données élémentaires, affectation, opérateurs arithmétiques | ||
| + | * Conditions : structures conditionnelles / expressions booléennes | ||
| + | * Itérations : structures itératives, types listes et chaînes de caractères | ||
| + | * Fonctions définies par le programmeur | ||
| + | * Gestion des entrées et sorties : interactif, lecture et écriture de fichiers, formats compressés<br> | ||
| + | |||
| + | Notions complémentaires | ||
| + | * Types structurés : listes, dictionnaires | ||
| + | * Expressions régulières (module re) | ||
| + | * Passage d’options et d’arguments à un script (sys, argparse) | ||
| + | * Modules : importation et création | ||
| + | * Modules spécifiques : biopython (bioinformatique), matplotlib (graphique), numpy (algèbre), sklearn (analyse de données, apprentissage automatique)<br> | ||
| + | |||
| + | Applications en bioinformatique | ||
| + | * Algorithmes de tri, de comptage, de recherche et de comparaison de séquences nucléiques et protéiques | ||
| + | * Transcription et traduction de séquences | ||
| + | * Lecture/écriture de fichiers FASTA et GenBank, extraction d’informations dans ce type de fichiers <br> | ||
| + | '''Références''' <br> | ||
| + | Apprendre à programmer avec Python 3, Gérard Swinnen, Eyrolles<br> | ||
| + | Programmation en Python pour les sciences de la vie, Patrick Fuchs, Pierre Poulain, DUNOD<br> | ||
| + | <!--competences=Maîtriser les notions fondamentales théoriques et pratiques d'un langage impératif | ||
| + | Savoir décomposer un problème en petits sous-problèmes et être capable d'implémenter une solution | ||
| + | Savoir comment évaluer la performance d'un programme|--> | ||
| + | '''Mots clefs''' : | ||
| + | Algorithmique ; structures ; instructions conditionnelles ; programmation impérative ; Biopython ; séquences génomiques nucléiques et protéiques ; annotations structurées ; | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Harmonisation des connaissances en biologie==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BBS''' <br> | ||
| + | '''ECTS : 3'''<br> | ||
| + | '''Niveau''' : Master 1 parcours BBS<br/> | ||
| + | '''Objectifs'''<br> | ||
| + | Cette UE est pour les étudiants ne provenant pas de biologie. Les étudiants venant de biologie suivront l'UE d'Algorithmique et complexité. L’objectif est de fournir un enseignement de mise à niveau en Biologie Cellulaire, Biologie Moléculaire et Génétique à des étudiants venant de licences en Physique ou en Informatique.<br> | ||
| + | '''Prérequis'''<br> | ||
| + | '''Contenu'''<br> | ||
| + | ''Biologie Cellulaire'' : Introduction générale à la biologie cellulaire. Notion de cellule procaryote et eucaryote. Organisation de la cellule (compartimentation et dynamique intracellulaire). Expression génique et régulation. Mouvements cellulaires. Prolifération, différenciation et mort cellulaire. Enseignement intéractif privilégiant un travail collectif. <br> | ||
| + | ''Analyse Génétique'' : Relation gène-fonction. Notions de mutation et d’allèle. Lois de transmission de caractères héréditaires chez les eucaryotes (i.e. : monogénique et digénique). Notions d’indépendance et de liaison génique. Transmission génétique chez les procaryotes. <br> | ||
| + | ''Biologie Moléculaire'' : Structure du matériel génétique, notions de gène et de génome. Présentation des grands processus moléculaires de la cellule (i.e. : réplication, transcription, traduction).<br> | ||
| + | '''Références''' <br> | ||
| + | Biologie (Campbell et Reece - De Boeck)<br> | ||
| + | competences=Niveau de base (licence) en Biologie Moléculaire, Biologie Cellulaire et Génétique --> | ||
| + | '''Mots Clefs''' : | ||
| + | Biologie cellulaire ; Biologie Moléculaire ; Génétique mendélienne et moléculaire | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Mathématiques pour la biologie==== | ||
| + | |||
| + | {{Template:Syllabus UE| | ||
| + | niveau=Master 1 parcours BBS| | ||
| + | ects=3| | ||
| + | objectifs=Initiation théorique aux bases d'algèbre linéaire, d'analyse et de probabilités. Représentation matricielle. Modélisation de problèmes dynamiques : résolution d'équations différentielles. Présentation des algorithmes numériques pour résoudre ces problèmes. Probabilités. Mise en application dans le cadre d’un projet applicatif en biologie des notions théoriques et algorithmes vus en cours.| | ||
| + | prerequis=| | ||
| + | contenu=''Algèbre Linéaire'' | ||
| + | * Opérations élémentaires sur les matrices | ||
| + | * Déterminant | ||
| + | * Diagonalisation : recherche de vecteurs/valeurs propres. | ||
| + | * résolutions de systèmes linéaires, | ||
| + | ''Analyse'' | ||
| + | * intégration, | ||
| + | * équations différentielles, | ||
| + | * optimisation, | ||
| + | * Droite de régression : problème des moindres carrés ordinaires | ||
| + | ''Probabilités'' | ||
| + | * Probabilités conditionnelles, règles de Bayes | ||
| + | * Variables aléatoires discrètes et continues : lois de probabilités, espérances, variances, covariance, corrélation | ||
| + | * modèle linéaire| | ||
| + | references=Mathématiques pour les Sciences de la vie et de la Terre – C. David, S. Mustapha, F. Viens, N. Capron, edition Dunod| | ||
| + | keywords=systèmes linéaires ; vecteurs/valeurs propres ; équations différentielles ; probabilités | ||
| + | }} | ||
| + | |||
| + | <!--competences=Savoir utiliser des outils mathématiques d'algèbre linéaire, d'analyse et de probabilités pour représenter et analyser des données biologiques, modéliser des problèmes dynamiques.--> | ||
| + | |||
| + | ====Traitement des données biologiques==== | ||
| + | {{Template:Syllabus UE| | ||
| + | niveau=Master 1 parcours BBS| | ||
| + | ects=3| | ||
| + | objectifs=Acquisition des notions fondamentales de l’analyse statistique appliquée aux données biologiques, et maîtrise de l’utilisation du logiciel R/Rstudio. Les étudiants doivent pouvoir (i) décrire des données biologiques associées à un plan expérimental, (ii) tester des hypothèses biologiques liées aux données par des méthodes statistiques adaptées, et (iii) interpréter biologiquement les résultats de ces traitements.| | ||
| + | prerequis=Connaissances de base en mathématiques. Notion d'expression génique.| | ||
| + | contenu=* Statistiques descriptives (une ou plusieurs variables qualitatives et/ou quantitatives, représentations graphiques, introduction aux analyses multivariées). | ||
| + | * Statistiques inférentielles (loi de probabilité, distribution d’échantillonnage, estimation ponctuelle, estimation par intervalle de confiance, tests paramétriques et non paramétriques, tests multiples). | ||
| + | * Introduction au traitement des données post-génomiques par l’analyse de données multivariées telles que des données de transcriptome : différence d’expression des gènes, recherche de co-régulations géniques par analyses multivariées et clustering, caractérisation d’ensembles de gènes.| | ||
| + | references=The R Book (Mickael J Crawley)| | ||
| + | keywords=statistiques descriptives ; probabilités ; échantillonnage ; test statistique ; analyses multivariées ; partitionnement | ||
| + | }} | ||
| + | |||
| + | <!--competences=Savoir décrire des données biologiques (incluant des données de transcriptome). Savoir tester des hypothèses liées aux données biologiques récoltées suite à l'experimentation/l'échantillonnage, et utiliser des approches statistiques courantes en sciences biologiques. Savoir interpréter biologiquement les résultats du traitement statistique. Maitriser un outil informatique appliqué à l’analyse statistique (logiciel R).--> | ||
| + | |||
| + | ====Bioinformatique des séquences==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BBS''' <br> | ||
| + | '''ECTS : 3'''<br> | ||
| + | '''Objectifs'''<br> | ||
| + | Comprendre les concepts et algorithmes sous-jacents aux principaux outils de comparaison de séquences biologiques afin d’être capable de choisir la méthode la plus pertinente pour répondre à une problématique. <br> | ||
| + | '''Prérequis'''<br> | ||
| + | Connaissance de biologie moléculaire et de bases en bioanalyse (contenu des banques, interrogation, recherche d'homologues). | ||
| + | '''Contenu''' <br> | ||
| + | Les cours viseront à montrer la différence entre un alignement exact (Needleman et Smith et Waterman) ou approché via des heuristiques (type BLAST) ; l'intérêt de la méthode de programmation dynamique pour la comparaison de séquences ; comprendre les différentes approches en alignement multiple de séquences : méthodes locales et globales, progressives versus itératives ; connaître les méthodes de caractérisation (signature, profile) et la recherche de motifs communs entre plusieurs séquences. | ||
| + | La mise en pratique de ces différentes méthodes sera faite lors de séances de travaux dirigés et pratiques, en insistant sur leurs avantages ou leurs limites.<br> | ||
| + | '''Références''' <br> | ||
| + | Bio-informatique. Principes d'utilisation des outils. 2010. D. Tagu, J.L. Risler, coord. Ed Quae <br> | ||
| + | Bioinformatique - Cours et applications. 2ème édition. 2015. G. Deléage et M. Gouy. Dunod <br> | ||
| + | <!--competences=Etre capable d'élaborer une stratégie et de choisir et utiliser les méthodes ad hoc pour comparer des ensembles de séquences et en dégager des informations pertinentes.--> | ||
| + | '''Mots clefs''' : Algorithmes pour la comparaison de séquences ; heuristique ; profils | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Génétique évolutive et quantitative==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BBS''' <br> | ||
| + | '''ECTS : 3'''<br> | ||
| + | '''Objectifs'''<br> | ||
| + | Acquisition des concepts fondamentaux de la génétique évolutive (génétique des populations) et de la génétique quantitative (architecture génétique des caractères quantitatifs, cartographie de QTL, association génotype/phénotype). Analyse de données génétiques et phénotypiques.<br> | ||
| + | '''Prérequis''' <br> | ||
| + | Notion de génétique Mendélienne (ségrégation des caractères) et de polymorphisme moléculaire.<br/> | ||
| + | '''Contenu''' <br> | ||
| + | * Génétique des populations : mesures du polymorphisme génétique, modèle de Hardy-Weinberg, systèmes de reproduction (autogamie, consanguinité, ...), forces évolutives (mutation, sélection, dérive génétique, migration). | ||
| + | * Génétique quantitative : modèle polygénique des caractères quantitatifs, modèle qualitatif, notion d’héritabilité, notion de ressemblance / apparentement génétique, principes de la cartographie de Quantitative Trait Loci (QTL) et de la génétique d’association. | ||
| + | *Ce contenu sera traité au travers d’exemples issus du monde végétal et de l’agronomie.<br> | ||
| + | '''Références''' <br> | ||
| + | Précis de génétique des populations (Jean-Pierre Henry, Pierre-Henry Gouyon, ed Dunond) <br> | ||
| + | Introduction to quantitative genetics (Falconer & Mackay) Handbook of statistical genetics (Balding, Bishop, Cannings)<br> | ||
| + | <!-- competences=Savoir mesurer le polymorphisme génétique des populations, et connaitre l'effet des systèmes de reproduction et des forces évolutives sur la diversité allélique et génotypique. | ||
| + | Savoir décrire un caractère quantitatif et évaluer l'importance des gènes et de l'apparentement dans la variabilité du caractère (héritabilité). | ||
| + | Connaitre le principe des approches de cartographie génétique des caractères quantitatifs (QTL, association).--> | ||
| + | '''Mots Clef'''s : génétique ; population ; évolution ; caractère quantitatif ; cartographie génétique | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Introduction aux bases de données==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BBS et Master 2 parcours BGE''' <br> | ||
| + | '''ECTS : 3'''<br> | ||
| + | '''Objectifs'''<br> | ||
| + | L'objectif de ce cours est d'apprendre aux étudiants à concevoir une base de données et de l’interroger en algèbre relationnelle ainsi qu'en langage déclaratif tel que le langage SQL.<br> | ||
| + | '''Prérequis''' <br> | ||
| + | '''Contenu''' <br> | ||
| + | # Définition, objectifs des bases de données et fonctions des SGBD | ||
| + | # Modèles de données | ||
| + | #* Modèle conceptuel : modèle Entité/Association E/A | ||
| + | #* Modèle logique : modèle relationnel | ||
| + | # Algèbre relationnelle | ||
| + | # Langages de définition et de manipulation des bases de données relationnelles | ||
| + | #* LDD : Langage de Définition de Données | ||
| + | #* LMD: Langage de Manipulation de Données <br> | ||
| + | '''Références''' <br> | ||
| + | Gardarin G., "Bases de données", Edition Eyrolles, 2003 (ISBN 2-212-11281-5).<br> | ||
| + | <!--competences=<br/> | ||
| + | * Maîtriser les concepts fondamentaux des bases de données relationnelles | ||
| + | * Savoir concevoir une base de données relationnelle | ||
| + | * Maîtriser le langage SQL --> | ||
| + | '''Mots Clefs''' : bases de données relationnelles ; modélisation ; algèbre relationnelle ; LDD ; LMD ; language SQL | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Bioinformatique pour la Génomique==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BBS et Master 2 parcours BGE''' <br> | ||
| + | '''ECTS : 3'''<br> | ||
| + | '''Objectifs'''<br> | ||
| + | Cet enseignement permettra aux étudiants d'acquérir les approches d'analyses des données de génomes, plus particulièrement l'annotation des séquences génomiques et la génomique comparative. Les concepts et les questions biologiques sous-jacents à ces approches seront abordés et seront suivis de leur mise en pratique sur des cas concrets.<br> | ||
| + | '''Prérequis''' <br> | ||
| + | '''Contenu'''<br> | ||
| + | Les cours aborderont : <br> | ||
| + | * La description de la conception d'un prédicteur de gène et des différentes méthodes qui doivent être mise en œuvre pour effectuer les mesures nécessaires sur la séquence génomique analysée (type contenu, signal, similarité) et comment ces différentes informations sont intégrées dans un modèle de structure de gène qui sera ensuite implémenté dans une solution logicielle permettant de réaliser la prédiction de la structure optimale. Parmi ces approches, les modèles de Markov cachés (HMM) permettant de réaliser des modèles probabilistes d’une suite de problèmes linéaires labellisés seront plus particulièrement développés. | ||
| + | * la description des concepts et des hypothèses fonctionnelles qui sous-tendent les approches de génomique comparative.<br> | ||
| + | Les séances de TP auront lieu sur ordinateur de manière à mettre en pratique les approches et démarches théoriques décrites en cours.<br> | ||
| + | '''Références''' <br> | ||
| + | Biological sequences analysis: Probabilistic Models of Proteins and Nucleic Acids (1998) R. Durbin, S. Eddy, A. Krogh, G. Mitchison. Cambridge University Press. <br> | ||
| + | '''Mots Clefs''' : Annotation de génomes ; Modèle de Markov caché ; génomique comparative ; alignement de génomes ; synténie | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Traitement de graphes et réseaux biologiques==== | ||
| + | |||
| + | {{Modèle:Syllabus_UE| | ||
| + | niveau=Master 1 parcours BBS et Master 2 parcours BGE| | ||
| + | ects=3| | ||
| + | objectifs=Dans ce module, les étudiants aborderont les concepts et les algorithmes de base en théorie des graphes. Quelques problèmes classiques de biologie seront revisités à la lumière de ces concepts. Les réseaux d’interactions moléculaires (régulation transcriptionnelle, réseaux métaboliques, réseaux d’interactions protéiques), les ontologies liées aux représentations des concepts biologiques, et les problèmes de graphes associés constitueront des applications privilégiées.| | ||
| + | prerequis=UE de programmation en bioinformatique| | ||
| + | contenu=* Concepts et définitions | ||
| + | * Graphes et réseaux rencontrés en biologie et bioinformatique | ||
| + | * Représentations informatiques et manipulations | ||
| + | * Formats de stockage | ||
| + | * Logiciels de visualisation, d'édition et d'analyse | ||
| + | * Librairies et boites à outils | ||
| + | * Algorithme de parcours et de dessin | ||
| + | * Arbres couvrants | ||
| + | * Détection de motifs | ||
| + | * Partitionnement et détection de communautés| | ||
| + | references=Introduction to Algorithms, Corsen, Leiserson and Rivest, MIT Press and McGraw-Hill| | ||
| + | keywords=parcours ; plus courts chemins ; marches aléatoires ; partitionnement de graphes ; détection de communautés ; modularité ; réseau d'interaction ; réseau de régulation ; réseau métabolique | ||
| + | }} | ||
| + | |||
| + | <!-- competences=Générer, éditer, manipuler et visualiser différents types de graphes. | ||
| + | Elaborer et mettre en œuvres des méthodes originales de parcours de réseaux biologiques. | ||
| + | Comparer les résultats de différentes méthodes de partitionnement de graphes, sélectionner les résultats biologiquement pertinents et interpréter les communautés obtenues.--> | ||
| + | |||
| + | ====Bioanalyse==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BGE''' <br> | ||
| + | '''ECTS : 3'''<br> | ||
| + | '''Objectifs'''<br> | ||
| + | Cet enseignement a pour but de former les étudiants aux approches bio-informatiques utilisées dans le cadre de l'analyse de séquences biologiques. Les concepts sous-jacents à ces approches seront décrits et seront suivis de leur mise en pratique.<br> | ||
| + | '''Prérequis'''<br> | ||
| + | Utilisation d'un navigateur web, notion de Biologie Moléculaire (ADN, ARN, protéines…)<br> | ||
| + | '''Contenu'''<br> | ||
| + | Les cours viseront en une présentation : des différentes banques de données et des systèmes d'interrogation ; des méthodes de recherche par similarité dans ces banques (Blast, Psi-Blast); des méthodes utilisées pour la comparaison de deux séquences (matrice de points, alignement global et local) ainsi que celles développées pour les alignements multiples; des matrices de substitution (PAM, BLOSUM etc..) ; des approches dédiées à la recherche de motifs, signatures et profils. | ||
| + | Les séances sur ordinateur illustreront les démarches et approches de bioanalyse décrites en cours, en utilisant des logiciels dédiés et des données biologiques disponibles dans les bases de données.<br> | ||
| + | '''Références''' <br> | ||
| + | Cours et applications (2015). Gilbert Deléage et Manolo Gouy. 2ème édition. DUNOD<br> | ||
| + | '''Mots Clefs''' : | ||
| + | Banque, BLAST, alignements, domaines et motifs. | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Analyse de données et modélisation en écologie==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BBS et BGE''' <br> | ||
| + | '''ECTS : 6''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | Maîtriser les méthodes d'analyse de données (outils statistiques et de modélisation) les plus communément utilisées pour l'analyse et la compréhension des phénomènes biologiques et écologiques. Faire le lien entre le questionnement biologique et les méthodes d'analyse à mettre en œuvre afin de maîtriser l'ensemble de la démarche expérimentale, depuis la formulation des hypothèses biologiques jusqu'à l'interprétation des résultats, en passant bien évidemment par l'analyse des données. Acquérir une bonne autonomie dans les analyses de données permettant de répondre à des problématiques écologiques simples mais variées.<br> | ||
| + | '''Prérequis''' <br> | ||
| + | Avoir déjà suivi un enseignement sur les probabilités, les statistiques descriptives et les tests d'hypothèses. Avoir des connaissances de base du logiciel R.<br> | ||
| + | '''Contenu''' <br> | ||
| + | Les concepts et méthodes d'analyse abordés concerneront les lois de probabilité, les tests d'hypothèses (paramétriques et non paramétriques), le modèle linéaire (plans expérimentaux, analyse de variance, régression simple et multiple), le modèle linéaire généralisé, la classification et l'analyse en composantes principales. Les aspects théoriques indispensables à la compréhension et à la bonne utilisation de ces méthodes seront traités au cours des CM. Les TP, qui auront lieu en salle informatique, permettront aux étudiants de mettre en pratique ces méthodes afin de répondre à des problématiques biologiques concrètes. Pour cela, ils seront familiarisés à un outil de traitement de données gratuit (le logiciel R). Une place importante sera donnée à l'interprétation écologique des résultats statistiques. Enfin, un projet réalisé en fin d'UE par binôme permettra aux étudiants d'être confrontés à un problème biologique concret. Ils devront ainsi identifier les méthodes d'analyse adaptées à la question biologique posée et acquérir les bons « réflexes » face aux difficultés de l'analyse de données en situation réelle.<br> | ||
| + | '''Références''' <br> | ||
| + | Bertrand F. & Maumy-Bertrand M. 2014. Initiation à la statistique avec R. Cours et exercices corrigés. Dunod.<br> | ||
| + | Scherrer B. 2008. Biostatistique volume 1. Editeur Gaëtan Morin.<br> | ||
| + | Crawley M.J. 2012. The R Book. Wiley.<br> | ||
| + | '''Mots Clefs''' : Analyses univariées et bivariées, modélisation, tests d'hypothèses, programmation, simulation. | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Ecologie évolutive==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BGE''' <br> | ||
| + | '''ECTS : 6''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | * Caractériser les variations (d'effectifs, de génotypes, de phénotypes et d'histoires de vie) dans les populations naturelles, | ||
| + | * Comprendre les mécanismes qui génèrent, maintiennent et modifient ces variations, | ||
| + | * Maîtriser les outils de description et d'analyse de ces variations<br> | ||
| + | '''Prérequis''' <br> | ||
| + | Maîtrise des concepts basiques en écologie, biologie évolutive, génétique, dynamique des populations, statistique inférentielle, mathématiques bac S.<br> | ||
| + | '''Contenu''' <br> | ||
| + | * Sélection naturelle (concepts et exemples) | ||
| + | * Dynamique des populations | ||
| + | * Génétique des populations (forces évolutives, structuration, régimes de reproduction, déséquilibre de liaison, coalescence) | ||
| + | * Dynamique adaptative (introduction) | ||
| + | * Génétique quantitative (héritabilité, équation du sélectionneur) | ||
| + | * Evolution des histoires de vie (mortalité, fécondité, itéroparité/semelparité, sénescence, âge à maturité, longévité…) | ||
| + | * Allocation au sexe de la descendance | ||
| + | * Dynamique et évolution des interactions (co-évolution, co-adaptation) | ||
| + | * Evolution expérimentale (expériences de terrain et en laboratoire, sélection artificielle)<br> | ||
| + | '''Références''' <br> | ||
| + | Thomas, F. et al. 2010. Biologie évolutive. Ed De Boeck<br> | ||
| + | Mayhew, P. J. 2006. Discovering Evolutionary Ecology: Bringing Together Ecology and Evolution. Oxford Univ. Press.<br> | ||
| + | Ricklefs, R. E., et Miller, G. L. 2005. Écologie. Ed De Boeck. <br> | ||
| + | '''Mots Clefs''' : Sélection, dynamique et génétique des pop., génétique quantit., dynamique adaptative, interactions, évolution expérimentale, histoire de vie, allocation au sexe. | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Ecologie des communautés et fonctionnement des écosystèmes==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BGE''' <br> | ||
| + | '''ECTS : 6''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | Les cours magistraux et les travaux dirigés apportent les bases conceptuelles et empiriques indispensables à la compréhension des règles d'assemblages des communautés et des forces régissant le fonctionnement des écosystèmes. L'écologie est présentée comme une discipline intégrative profondément enracinée dans les sciences naturelles, mais dont les bases théoriques dérivent de principes généraux issus de la biologie, de la physique et de la chimie. Les enseignements apportent aussi un éclairage sur l'écologie en tant que science appliquée au service de l'humanité.<br> | ||
| + | '''Prérequis''' <br> | ||
| + | Connaissances académiques en biologie des organismes et des populations. Compréhension de la démarche scientifique et des méthodes d'analyse de données.<br> | ||
| + | '''Contenu''' <br> | ||
| + | ''Cours'' : <br> | ||
| + | * Les multiples facettes de la biodiversité et leurs relations | ||
| + | * Le concept de niche écologique : émergence, évolution, et applications | ||
| + | * Règles d'assemblage et de dé-sassemblage des communautés | ||
| + | * Dynamique des réseaux d'interactions | ||
| + | * Evolution et stabilité des écosystèmes | ||
| + | * Cycle de la matière organique; cycles des éléments et stoechiomètrie écologique | ||
| + | * Rôle de la biodiversité dans le fonctionnement des écosystèmes et les services écologiques | ||
| + | * Influence de la structure de l'habitat, des facteurs physico-chimiques et des communautés dans le fonctionnement des sols et des systèmes aquatiques | ||
| + | * Ecologie appliquée : restauration et ingénierie écologique | ||
| + | * L'écologie à l'échelle du paysage et des méta-écosystèmes | ||
| + | ''Travaux dirigés'' : | ||
| + | * Outils numériques pour l'étude de la biodiversité et la modélisation des écosystèmes | ||
| + | * Utilisation des biomarqueurs et des bioindicateurs en écologie<br> | ||
| + | '''Références''' <br> | ||
| + | Weathers K.C. et al. (2013) Fundamentals of Ecosystem Science. Academic Press <br> | ||
| + | Morin P.J. (2011) Community Ecology, Wiley-Blackwell <br> | ||
| + | Naeem S. et al. (2009) Biodiversity, Ecosystem Functioning, & Human Wellbeing: Oxford University Press <br> | ||
| + | '''Mots Clefs''' : biodiversité, écosystème, autoécologie, synécologie, traits fonctionnels, cycle de la matière organique et des nutriments, ingénierie écologique. | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Programmation avancée et génie logiciel==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BBS''' <br> | ||
| + | '''ECTS : 6''' <br> | ||
| + | '''Objectifs''' | ||
| + | * Maitriser la modélisation objets d’un programme informatique | ||
| + | * Connaitre les différents concepts de la programmation orientée objet | ||
| + | * Savoir développer une application en utilisant une approche orientée objet | ||
| + | * Maitriser l’utilisation d’API dans le développement d’un programme informatique | ||
| + | * Connaitre les principaux éléments permettant de concevoir et développer une interface graphique <br> | ||
| + | '''Prérequis''' | ||
| + | * Bonne connaissance des base de l’algorithmique | ||
| + | * Première expérience avec un langage de programmation| | ||
| + | '''Contenu''' <br> | ||
| + | ''Partie génie logiciel : Modélisation avec UML'' | ||
| + | * Introduction sur l'intérêt de la modélisation | ||
| + | * Présentation des cas d'utilisation et des principaux diagrammes (diagramme de classe, de séquence, d’état transition) | ||
| + | ''Partie programmation orientée objet - Langage JAVA'' | ||
| + | * Introduction du langage JAVA | ||
| + | * Présentation des différents outils de compilation, éxecution, documentation, etc. et des principaux environnements de programmation (Eclipse, NetBeans) | ||
| + | * Notion de classe et objet | ||
| + | ** Principales caractéristiques d'une classe (notion d'attributs, constructeurs, méthodes, visibilité) | ||
| + | ** Instanciation des classes, | ||
| + | ** Utilisation de package | ||
| + | ** Lien avec la modélisation UML | ||
| + | * Notion avancée de programmation orientée objet : héritage, interface, classe abstraite | ||
| + | * Gestion des exceptions | ||
| + | * Présentation d'API | ||
| + | ** Différentes structures de données | ||
| + | ** Gestion des flux de données : utilisation de fichiers, lecture et écriture dans des flux, sérialisation, parseur XML | ||
| + | ** Introduction aux interfaces graphiques et à la programmation événementielle <br> | ||
| + | '''Références''' <br> | ||
| + | <!--competences=Être capable de modéliser et développer une application complète (avec, par exemple, la gestion de la lecture et écriture dans des fichiers, et la conception d'une interface graphique) en utilisant la programmation orientée objet.--> | ||
| + | '''Mots Clefs''' : Modélisation objet ; UML ; Programmation orientée objet ; JAVA ; Classes ; Objets ; Héritage | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Génomique Environnementale==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BGE''' <br> | ||
| + | '''ECTS : 6''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | * Mobiliser les concepts fondamentaux relatifs à la génomique environnementale, les objets qu’elle permet d’étudier (communauté complexe, génome d’organismes non modèles, niveau de variabilité, ADN ancien, détection d’espèces rares), les conditions de mise en oeuvre (échantillonnage, choix des marqueurs et des techniques de séquençage), les outils bioinformatiques nécéssaires (différents pipelines), les limitations et précautions (variabilité intra et interspécifique, contaminations, seuils…) | ||
| + | * Identifier, choisir une combinaison de méthodes et d’outils analytiques relatifs aux NGS pour répondre à des questions d’écologie et d’évolution. | ||
| + | * Interpréter des résultats issus de l’analyse de données de génomique environnementale - qu’ils soient originaires d’un article ou des analyses menées par les étudiants. | ||
| + | * Exploiter des logiciels d’acquisition et d’analyse de données NGS avec un esprit critique.<br> | ||
| + | '''Prérequis''' <br> | ||
| + | '''Contenu''' <br> | ||
| + | Les ''cours magistraux'' permettront d’introduire la génomique environnementale, ses questions, ses objets, ses technologies, puis d’explorer trois grands thèmes autour des usages de la génomique environnementale pour étudier (1) l’ADN ancien ou dégradé, (2) l’écologie microbienne et les interactions, et enfin (3) la comparaison des génomes, tant d’eucaryotes que de procaryotes.<br> | ||
| + | Les ''TD'' seront dédiés à la présentation de cas d’étude sous forme d’article, pour pouvoir décrypter l’ensemble des étapes d’un projet de génomique environnementale, avec un oeil critique. Un des TD sera aussi dédié à une visite virtuelle ou non d’une plateforme de séquençage. L’application de ces techniques dans le domaine de l’écologie de la conservation sera aussi abordée dans le cadre des TD.<br> | ||
| + | Enfin les ''TP'' aborderont deux des trois grands thèmes présentés, et permettront d’appliquer des outils bioinformatiques, et décrypter les lignes de commandes, et d’aboutir à des résultats que les étudiants devront interpréter.<br> | ||
| + | '''Références''' <br> | ||
| + | '''Mots Clefs''' <br> | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Fouille de données==== | ||
| + | {{Template:Syllabus UE| | ||
| + | niveau=Master 1 parcours BBS et BGE| | ||
| + | ects=3| | ||
| + | objectifs=Cette UE a pour but d’initier les étudiants aux techniques modernes de fouilles de données permettant de prédire, par exemple, si celui qui achète du pain et du beurre va acheter de la confiture, s’il est raisonnable pour une banque d'attribuer une carte de crédit, si la protéase PfSUB1 joue un rôle dans la division de ''Plasmodium falciparum'' (parasite responsable de la malaria), ou encore de diagnostiquer un sous-type de cancer du sein à partir de données transcriptomique de la patiente. En général, plusieurs méthodes peuvent être utilisées, plus ou moins performantes : il s’agit donc de les comprendre, d’apprendre à les mettre en œuvre et d’estimer leurs performances.| | ||
| + | prerequis=Statistiques, programmation| | ||
| + | contenu=*Mesures de distance, similarité, dissemblance | ||
| + | *Normalisation et transformation | ||
| + | *Classification | ||
| + | **Arbre de décision et forêts aléatoires | ||
| + | **Méthodes bayésiennes | ||
| + | **k plus proches voisins | ||
| + | **Analyse discriminante linéaire | ||
| + | *Clustering | ||
| + | **k means, k medoids | ||
| + | **Clustering hiérarchique | ||
| + | **Méthodes basées sur la densité | ||
| + | **Méthodes basées sur une grille | ||
| + | *Evaluation des performances | ||
| + | *Extraction de règles d'association et d'itemsets fréquents | ||
| + | *Logiciels, librairies et boites à outils disponibles| | ||
| + | keywords=prétraitement des données ; classification et prédiction ; partitionnement ; caractérisation et discrimination ; règles d’association| | ||
| + | references=Data Mining: Concepts and Techniques, J. Han and M. Kamber, 2006<br/> | ||
| + | Hadley Wickham & Garret Grolemund, ''R for Data Science'', https://r4ds.had.co.nz <br/> | ||
| + | Pedregosa ''et al.'', ''Scikit-learn: Machine Learning in Python'', JMLR, 2011 https://scikit-learn.org/ | ||
| + | }} | ||
| + | |||
| + | <!-- competences=Constituer un jeu de données d'apprentissage pour l'inférence d'un modèle prédictif. | ||
| + | Evaluer les performances d'une méthode de classification, d'un classificateur, ou d'un jeu de paramètres. | ||
| + | Sélectionner et mettre en œuvre une méthode de clustering ainsi que évaluer et interpréter le résultat obtenu--> | ||
| + | |||
| + | ====Traitement de données post-génomiques==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BBS et BGE''' <br> | ||
| + | '''ECTS : 6''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | Cette unité d'enseignement a pour objectif de fournir une large palette de connaissances et de compétences en bioinformatiques et biostatistiques appliquées à un domaine en constante évolution en biologie moderne: la post-génomique. Cette dernière cherche à étudier la fonction et l'expression des gènes séquencés en génomique par la caractérisation systématique du rôle des mutations, de l'expression des ARN messagers, des ARN non-codants, ainsi que des protéines. Les données générées par la post-génomique sont extrêmement volumineuses et de très grandes dimensions (Big Data). Les applications sont nombreuses: génétique humaine, cancérologie, métagénomique, mais aussi, amélioration des espèces d'intérêt agronomique et étude de l'évolution des espèces.<br> | ||
| + | '''Prérequis''' <br> | ||
| + | Connaissances en biologie moléculaire, génomique, biochimie des protéines, ainsi qu'en algorithmique, théorie des graphes et traitement statistique de données.<br> | ||
| + | '''Contenu''' <br> | ||
| + | Le premier atelier aborde la manipulation de gros fichiers de séquences, l'alignement des séquences sur le génome, l'assemblage de novo de génomes, la détection de SNP ainsi que leur annotation fonctionnelle. Le second atelier traite de la mesure du niveau d'expression des ARN messagers, de l'analyse différentielle, des méthodes d'enrichissement d'ensemble de gènes et de la détection de variants d'épissage alternatif. Le troisième atelier permet de caractériser les différentes familles d'ARN non codants, d'employer les banques de données et de maîtriser les méthodes bioinformatiques pour traiter les données. Le quatrième atelier présente les caractéristiques générales des spectromètres de masse et les stratégies d'analyse protéomique. Il traite aussi de l'analyse des modifications post-traductionnelles et à celle des interactomes, des stratégies de quantification haut-débit et du traitement des données protéomiques.<br> | ||
| + | '''Références''' <br> | ||
| + | Pevsner. Bioinformatics and Functional Genomics, 2015. <br> | ||
| + | Datta and Nettelton. Statistical Analysis of Next Generation Sequencing Data, 2014. <br> | ||
| + | Wu and Choudry, Next Generation Sequencing in Cancer Research, 2013.<br> | ||
| + | '''Mots Clefs''' : Haut-débit ; Assemblage de génome ; SNP ; RNA-seq ; small RNA-seq ; Protéomique | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Evolution moléculaire==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BBS et BGE''' <br> | ||
| + | '''ECTS : 3''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | Cet enseignement a pour but d'initier les étudiants aux analyses d'évolution moléculaire. Les concepts et méthodes développées dans cette discipline seront détaillés et seront accompagnés par leur mise en pratique sur des cas concrets de reconstruction phylogénétique. L'accent sera mis sur l'interprétation évolutive des arbres obtenus. <br> | ||
| + | '''Prérequis''' <br> | ||
| + | '''Contenu''' <br> | ||
| + | Cette UE introduira les concepts de l’évolution puis présentera les différents modèles d'évolution des séquences génomiques (ADN et protéines) et les différentes approches de reconstruction d’arbres (parcimonie, méthode de distances, méthode du maximum de vraisemblance). Les méthodes permettant le choix du modèle évolutif le plus adapté aux données traitées, celles utilisées pour analyser la stabilité de la topologie et celles permettant l'analyse de la congruence de plusieurs arbres seront également développées. L'étude de l'impact des forces évolutives (sélection naturelle, dérive,…) sur le polymorphisme des séquences sera aussi abordé. Les concepts et approches vus en cours seront illustrés par des cas concrets (évolution des séquences d'une famille de protéines, pression de sélection sur certains gènes et régions du génome, reconstruction de la phylogénie d'un ensemble d'espèces, etc) lors de séances de TP sur ordinateurs. Au cours des TD, l'accent sera mis sur l'interprétation évolutive des topologies d'arbres (détection des évènements de duplication et/ou perte de gène, de transferts horizontaux etc.)<br> | ||
| + | '''Références''' <br> | ||
| + | Concepts et Méthodes en Phylogénie Moléculaire (2010). Guy Perrière et Céline Brochier-Armanet. Collection IRIS, Springer <br> | ||
| + | Computational Molecular Evolution. Ziheng Yang. Oxford University Press.<br> | ||
| + | <!-- competences=Mettre en œuvre une analyse évolutive des séquences d'une famille de gènes/protéines en utilisant les bonnes pratiques du choix du modèle évolutif le plus adapté aux données et des méthodes de reconstruction d'arbres. | ||
| + | Evaluer les différents types de sélection et leur impact sur le polymorphisme des séquences, et mettre en oeuvre une méthode de recherche de traces de sélection (tests de neutralité). | ||
| + | Interpréter un arbre phylogénétique pour proposer un scénario évolutif des séquences analysées.--> | ||
| + | '''Mots Clefs''' : évolution ; méthodes de reconstruction d’arbre phylogénétique ; congruence ; bootstrap ; sélection naturelle ; dérive | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Projet tutoré en bioinformatique==== | ||
| + | |||
| + | {{Template:Syllabus UE| | ||
| + | niveau=Master 1 parcours BBS et BGE| | ||
| + | ects=3| | ||
| + | prerequis=UE du premier semestre du M1| | ||
| + | objectifs=Mettre en œuvre des techniques de méthodologie de travail et d'organisation de projet. | | ||
| + | contenu=''Partie I''<br/> | ||
| + | Méthodologie de travail en groupe et de suivi de projet. Outils de suivi de modifications et de travail collaboratif. Licences logicielles. <br/> | ||
| + | ''Partie II'' <br/> | ||
| + | Projets en groupes de 3 à 4 personnes. Travail sur des thématiques de bioinformatique mettant en œuvre les concepts et compétences acquises au premier semestre. Utilisation de techniques de travail en groupe et de suivi de projets. Évaluation par des rapports de suivi de projet et une soutenance orale.| | ||
| + | references=PMI, management de projet : un référentiel de connaissances, AFNOR, 2004 <br> | ||
| + | Management d'un projet système d'information : principes, techniques, mise en œuvre et outils, Chantal Morley, Dunod, 2006| | ||
| + | keywords=méthodologie de travail ; travail en groupe ; suivi de projet ; suivi des modifications ; outils collaboratifs | ||
| + | }} | ||
| + | |||
| + | <!--competences=maîtrise des techniques de management des projets informatiques dans le domaine de la bioinformatique--> | ||
| + | |||
| + | ====Introduction Modélisation Moléculaire==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BBS''' <br> | ||
| + | '''ECTS : 3''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | Ce module a pour but d'initier les étudiants aux concepts de base de la modélisation moléculaire. A l'issu de cet enseignement l'étudiant sera capable de créer une molécule et de déterminer sa structure optimale, tant in vacuo que dans un complexe récepteur-ligand.<br> | ||
| + | '''Prérequis''' <br> | ||
| + | '''Contenu''' <br> | ||
| + | Présentation de concepts de base de la modélisation de structures biomoléculaires. Seront abordés les aspects théoriques et computationnels de la détermination de structures tridimensionnelles de molécules d'intérêt biologique par l'approche empirique, basée sur le champ de force et l'optimisation de géométrie. La partie pratique du module sera consacrée à la création, visualisation, modification et optimisation de structures moléculaires. <br> | ||
| + | '''Références''' <br> | ||
| + | Molecular Modelling: Principles and Applications", Andrew Leach, Prentice Hall, 2001.<br> | ||
| + | "Molecular Modelling for Beginners", Alan Hinchliffe, John Wiley & Sons Ltd, 2003.<br> | ||
| + | "Molecular Modeling and Simulation", Tamar Schlick, Springer-Verlag New York Inc., 2010.<br> | ||
| + | <!-- competences=Savoir créer une molécule et savoir déterminer sa structure optimale, tant in vacuo que dans un complexe récepteur-ligand.--> | ||
| + | '''Mots Clefs''' : Visualisation et manipulation de molécules ; structure et dynamique moléculaire ; champs de force ; minimisation d'énergie ; complexe récepteur-ligand | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Analyse de données multivariées==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BBS et BGE''' <br> | ||
| + | '''ECTS : 3''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | A l'issue de cet enseignement, les étudiants devront être à même : | ||
| + | * d'organiser des données et de formuler une problématique pertinente | ||
| + | * de choisir la (ou les) méthode(s) d'analyse en fonction de la nature des données et de la problématique formulée | ||
| + | * de mettre en œuvre ces méthodes (utilisation du logiciel R) | ||
| + | * de représenter graphiquement et d'interpréter les résultats | ||
| + | * de rédiger les conclusions dans un rapport de synthèse| | ||
| + | '''Prérequis''' <br> | ||
| + | Une UE de biostatistiques élémentaires est exigée, ainsi que des connaissances de base du logiciel R.<br> | ||
| + | '''Contenu''' <br> | ||
| + | Cet enseignement propose une présentation des principales méthodes d'analyse adaptées aux données multidimensionnelles. Les méthodes abordées seront illustrées à partir d'exemples réels provenant d'études écologiques. L'enseignement cherchera à montrer plus particulièrement en quoi (1) la nature complexe des systèmes biologiques conduit souvent à la nécessité de prendre en compte un grand nombre de descripteurs, et (2) l'écologie est un champ d'application privilégié des diverses méthodes abordées. <br> | ||
| + | Les aspects théoriques indispensables à la compréhension et à la bonne utilisation de ces méthodes seront traités au cours des CM. Les TP, en salle informatique, permettront aux étudiants de mettre en pratique ces méthodes et donner une place importante à l'interprétation écologique des résultats statistiques. Enfin, un projet réalisé en fin d'UE par binôme permettra aux étudiants d'être confrontés à un problème biologique concret. Ce projet portera sur l'analyse d'un jeu de données collecté dans le cadre d'une étude écologique et aboutira à la rédaction d'un rapport et une présentation orale du travail effectué.<br> | ||
| + | '''Références''' <br> | ||
| + | Escoffier & Pagès. Analyses factorielles simples et multiples – Objectifs, méthodes et interprétation. Dunod <br> | ||
| + | Lebart, Piron, & Morineau. Statistiques exploratoire multidimensionnelle. Dunod<br> | ||
| + | '''Mots Clefs :''' Analyses multivariées ; structure des tableaux de données en écologie ; liaisons entre descripteurs (biologiques, environnementaux, ...). | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Biostatistiques : Utilisation avancée du modèle linéaire==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BBS et BGE''' <br> | ||
| + | '''ECTS : 3''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | A l'issue de cet enseignement, les étudiants devront être à même d'analyser un jeu de données complexe en utilisant des modèles linéaires (lm) ou le des modèles linéaires généralisés (glm). Ils devront maîtriser suffisamment les bases théoriques de ces outils pour pouvoir montrer la pertinence de leur choix d'analyse, et interpréter en détail les résultats obtenus.<br> | ||
| + | '''Prérequis''' <br> | ||
| + | Statistiques descriptives, lois de probabilités usuelles, test d'hypothèse, régression linéaire, ANOVA, ANCOVA, utilisation basique du logiciel R.<br> | ||
| + | '''Contenu''' <br> | ||
| + | Cet enseignement propose une présentation détaillée des applications du modèle linéaire et du modèle linéaire généralisé à l'analyse des données biologiques. Les éléments théoriques permettant de comprendre les conditions d'application de ces méthodes d'analyse seront expliqués. L'accent sera mis sur les outils permettant de vérifier que ces conditions d'application sont bien remplies, et sur la démarche à adopter lorsqu'elles ne le sont pas.<br> | ||
| + | L'enseignement sera illustré en travaux pratiques par l'analyse détaillée de jeux de données tirés de travaux en écologie, biologie comportementale et biologie évolutive.<br> | ||
| + | '''Références''' <br> | ||
| + | The R Book, 2nd Edition. M.J. Crawley 2012. Wiley<br> | ||
| + | '''Mots Clefs''' : Statistique ; Modèle linéaire ; Modèle linéaire généralisé | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Stage (facultatif)==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 1 parcours BBS et BGE''' <br> | ||
| + | '''ECTS : 0''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | Ce stage optionnel permet aux étudiants d'avoir un premier contact avec le monde professionnel et donc une première immersion dans une équipe de recherche et développement appartenant soit à un laboratoire du milieu académique ou d'une entreprise. <br> | ||
| + | '''Prérequis''' <br> | ||
| + | '''Contenu''' <br> | ||
| + | Un stage optionnel d'un à deux mois est proposé en fin d'année universitaire car le besoin d'acquérir et/ou de renforcer des compétences dans différents champs disciplinaires (informatique, mathématiques et bioinformatique) ne permet pas d'introduire un stage obligatoire dans le cursus. Il est cependant fortement conseillé d'effectuer ce stage car il permettra d'apprendre à travailler en équipe, d'organiser son travail de manière à répondre aux échéances exigées par l'encadrant, de trouver des solutions pour résoudre des problèmes qui n'avaient pas été envisagés au départ. C'est également l'opportunité de confronter sa formation académique à des problématiques concrètes et de de mettre en oeuvre les compétences acquises. Il constitue une bonne préparation au stage obligatoire de 6 mois de la seconde année de master.<br> | ||
| + | '''Mots Clefs''' : Immersion professionnelle | ||
| + | </font> | ||
| + | |||
| + | === UE Master 2 === | ||
| + | ====Bases de données avancées==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 2 parcours BBS''' <br> | ||
| + | '''ECTS : 3''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | Cet enseignement a pour objectif de former les étudiants aux concepts avancés liés aux bases de données relationnelles : évaluation d'une requête relationnelle, interrogation d'une base de données via un langage procédural, administration d'une base de données. <br> | ||
| + | '''Prérequis''' <br> | ||
| + | Les concepts fondamentaux en bases de données (modèles entité/association, modèle relationnel, notions fondamentales en SQL)<br> | ||
| + | '''Contenu''' <br> | ||
| + | D'abord, les différentes étapes pour l'évaluation d'une requête relationnelle dans un environnement centralisé sont passées en revue. Ensuite, une introduction à un langage procédural est faite afin d'interroger et d’administrer une base de données.<br> | ||
| + | '''Références''' <br> | ||
| + | Georges Gardarin - Bases de Données - EYROLLES <br> | ||
| + | '''Mots Clefs''' : Bases de données ; Système de gestion de bases de données ; PL/SQL; Vues relationnelles. | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Introduction Apprentissage automatique==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 2 parcours BBS et BGE''' <br> | ||
| + | '''ECTS : 3''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | L'apprentissage automatique ou apprentissage artificiel est un champ d'étude de l'intelligence artificielle qui se fonde sur des approches mathématiques et statistiques pour donner aux ordinateurs la capacité d'« apprendre » à partir de données, c'est-à-dire d'améliorer leurs performances à résoudre des tâches sans être explicitement programmés pour chacune. Plus largement, il concerne la conception, l'analyse, l'optimisation, le développement et l'implémentation de telles méthodes. Les progrès récents de ce champ de l'IA font qu'il s'introduit dans tous les champs scientifiques, et en particulier la biologie et la génétique. | ||
| + | Le but du module est donc de donner aux étudiant(e)s un aperçu global de l'apprentissage, à la fois l'apprentissage par renforcement et l'apprentissage à partir d'exemples. Pour ces 2 sous domaines, les principes théoriques sont introduits, des algorithmes sont présentés. La partie pratique consiste en l'implémentation en python de ces algorithmes ainsi que la manipulation des librairies essentielles au domaine (Keras, tensorflow, scikit-learn). | ||
| + | A la fin du module, les étudiant(e)s sont capables d'analyser un jeu de données, de le préparer, de choisir un algorithme d'apprentissage adapté et de l'implémenter.<br> | ||
| + | '''Prérequis''' <br> | ||
| + | Calcul différentiel; Probabilités (les fondamentaux seulement) <br> | ||
| + | '''Contenu''' <br> | ||
| + | ''Apprentissage par renforcement'' | ||
| + | * Définition | ||
| + | * Equation de Bellman | ||
| + | * Q-table – implémentation en python | ||
| + | ''Apprentissage à partir d’exemples'' | ||
| + | * Principes génériques et fonction de perte | ||
| + | * Minimisation et descente en gradient | ||
| + | * Réseau de neurones | ||
| + | * Re-implémentation de la Q-table via réseau de neurones | ||
| + | * Convolution pour traitement d’images | ||
| + | * Problèmes et solutions : vanishing gradient, overfitting, etc. | ||
| + | * SVM | ||
| + | '''Références''' <br> | ||
| + | Machine learning (1997) Tom M. Mitchel, Publisher: McGraw-Hill <br> | ||
| + | '''Mots Clefs''' : Apprentissage automatique ; Règles d’association ; Réseau de neurones multi-couches | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Gestion de données non structurées et applications post-génomiques==== | ||
| + | |||
| + | {{Template:Syllabus UE| | ||
| + | niveau=Master 2 parcours BBS| | ||
| + | ects=3| | ||
| + | objectifs=Ce module a pour but d'enseigner aux étudiant·e·s comment modéliser, représenter et exploiter les masses de données complexes et variées disponibles dans les données publiques et/ou issues des approches de post-génomiques.| | ||
| + | prerequis=mathématiques ; statistiques ; traitement de graphes ; fouille de données ; bases de données relationnelles| | ||
| + | contenu=Introduction aux systèmes de gestion de données non structurées (NoSQL).<br/> | ||
| + | Modélisation, stockage et interrogation de données selon plusieurs modèles NoSQL (orienté documents et orienté graphe).<br/> | ||
| + | Alimentation de la base de données par des sources de données hétérogènes.<br/> | ||
| + | Exploitation de cette base par des exemples de projets de post-génomique comme par exemple la caractérisation d'un ensemble de gènes ou la priorisation de gènes candidats.| | ||
| + | references=Christian Theil Have, Lars Juhl Jensen, Are graph databases ready for bioinformatics?, Bioinformatics, Volume 29, Issue 24, 15 December 2013, Pages 3107–3108 https://doi.org/10.1093/bioinformatics/btt549 <br/> | ||
| + | Polyane Wercelens et al., Bioinformatics Workflows With NoSQL Database in Cloud Computing, Evol Bioinform Online, 2019<br/> | ||
| + | Les bases de données NoSQL et le BigData : Comprendre et mettre en oeuvre Ed. 2. Auteur: Bruchez, Rudi, Eyrolles (2015). ISBN: 978-2-212-14155-9<br/> | ||
| + | Bases de données orientées graphes avec Neo4j - Manipuler et exploiter vos bases de données orientées graphes. Auteurs: Benhenni, Amine Lies,Bois, François-Xavier. Eyrolles (2016). ISBN: 978-2-212-13804-7| | ||
| + | keywords=masses de données non structurées ; données hétérogènes ; caractérisation d'un ensemble | ||
| + | }} | ||
| + | |||
| + | ====Phylogénomique==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 2 parcours BBS et BGE''' <br> | ||
| + | '''ECTS : 3''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | La phylogénomique est une nouvelle discipline à l’intersection de l’évolution moléculaire et de la génomique. Elle permet de reconstruire l’histoire évolutive des espèces au travers de leurs génomes et ainsi de fournir un cadre conceptuel dans lequel l’évolution des gènes et des organismes peut être étudiée. L’identification des gènes orthologues entre espèces contribue à la prédiction de la fonction des gènes par transferts d’annotation. L’analyse des répertoires de gènes d’organismes se développant dans des environnements différents permet d’identifier les gènes impliqués dans l’adaptation à des niches écologiques. Cette approche a une contribution majeure pour comprendre la mise en place de résistances aux antibiotiques et l’adaptation des bactéries à leurs hôtes. Elle est en plein essor avec la prolifération des données issues de l’environnement et du milieu médical et grâce aux progrès réalisés pour analyser des gros volumes de données par des méthodes bioinformatiques. Le but de cet enseignement est d’initier les étudiant(e)s aux approches phylogénomiques en détaillant les principes théoriques des méthodes impliquées dans les différentes étapes de l’analyse. La partie pratique consistera à développer une chaîne de traitements en s’appuyant sur une question biologique particulière utilisant des données « réelles » issues de la littérature. A l’issue de cette UE, les étudiant(e)s sont capables de conduire un même type d’analyse sur des données différentes. <br> | ||
| + | '''Prérequis''' <br> | ||
| + | Les UE d’Evolution Moléculaire, de Bioinformatique pour la Génomique et de Traitement de graphes et réseaux biologiques. <br> | ||
| + | '''Contenu''' <br> | ||
| + | Les différentes approches, de la théorie à la pratique, seront introduites au travers de cours. La mise en œuvre se fera dans le cadre de séances de travaux pratiques utilisant un cluster de calcul. | ||
| + | Les points suivant seront abordés : | ||
| + | * Construction du jeu de données en fonction des objectifs avec une sélection des génomes sur la base de leur qualité (complétude et contamination) et de leur représentativité (réduction de la redondance et optimisation de la diversité). | ||
| + | * Evolution de la structure des génomes (duplication, délétions, inversions, transposition…) par alignements de génomes et par des méthodes basées sur des graphes. | ||
| + | * Evolution du répertoire des gènes (duplication, délétion, recombinaison, conversion, transfert de gènes) et identification des liens d’homologies, d’orthologies et de paralogies. | ||
| + | * Construction de groupes de gènes orthologues entre deux et plus de deux génomes. | ||
| + | * Identification du génome cœur, génome accessoire, et pangénome pour une espèces représentée par plusieurs souches. | ||
| + | * Construction d’un arbre espèce par différentes approches. | ||
| + | * Utilisation de cet arbre pour réconcilier les arbres gènes pour identifier les duplications, pertes et transferts de gènes. | ||
| + | * Utilisation de cet arbre pour la reconstruction d’états ancestraux pour des données discrètes et continues.<br> | ||
| + | Les TP permettront de mettre en pratique les approches et démarches théoriques décrites en cours sur des données « réelles » issues de la littérature. Au cours de ces TP, les étudiants construiront une chaîne de traitements pour automatiser les différentes étapes d’une analyse phylogénomique. Comme exemples de modèles d’études, nous pouvons citer (i) l’étude de l’évolution des génomes des espèces de ''Prochlorococcus'' (un genre de cyanobactéries marines photosynthétiques) en lien avec l’adaptation des différents clades de ''Prochlorococcus'' à différentes niches écologiques et (ii) l’étude de l’évolution des génomes des différentes souches de ''Staphylococcus aureus'' en lien avec leur capacité à franchir la barrière d’espèce et à acquérir des résistances aux antibiotiques. | ||
| + | '''Références''' <br> | ||
| + | '''Mots Clefs''': phylogénomique ; évolution ; orthologie ; génomique comparative''' <br> | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Métagénomique écologique et évolutive==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 2 parcours BGE''' <br> | ||
| + | '''ECTS : 9''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | ''S’approprier'' les approches : | ||
| + | * de génomique des populations qui permettent d’évaluer l’influence des pressions évolutives (sélection naturelle, dérive génétique, recombinaison, mutation, et migration) sur la diversité génétique d’une espèce. | ||
| + | * de génomique écologique, qui permettent d’identifier les bases génétiques (i) des caractères adaptatifs et (ii) de la réponse des populations à la variabilité de l’environnement biotique et abiotique. | ||
| + | * de métagénomique qui permettent de décrire la diversité taxonomique et fonctionnelle, de modéliser les interactions entre les différentes composantes, et d’émettre des hypothèses sur leur évolution et leur capacité d’adaptation dans un contexte d’environnement changeant. | ||
| + | * de paléogénomique qui permettent de comprendre l’évolution démographique et adaptative de certains groupes taxonomiques sous un angle temporel. <br> | ||
| + | ''Exploiter'' des données génomiques de polymorphismes moléculaires provenant d’échantillons populationnels ou environnementaux afin de tester des hypothèses dans le cadre de ces approches, par l’utilisation de logiciels de modélisation (méthodes statistiques descriptives et inférentielles). <br> | ||
| + | ''Interpréter'' les résultats issus de l’analyse de données génomiques populationnelles ou environnementales - qu’ils soient originaires d’un article ou des analyses menées par les étudiants.<br> | ||
| + | '''Prérequis''' <br> | ||
| + | Les UE de M1 BGE : Génomique Environnementale, Ecologie Evolutive <br> | ||
| + | '''Contenu''' <br> | ||
| + | Les différentes approches, de la théorie à la pratique, seront introduites au travers de CM, séminaires et TD de chercheurs et d’enseignants-chercheurs, en s’appuyant sur des projets de recherche existants. Les TD pourront s’appuyer sur l’analyse d’articles méthodologiques ou de recherche ciblant certaines thématiques. La mise en application sur les 4 grandes thématiques se fera dans le cadre de séances de travaux pratiques et de mini-projets, par l’analyse et l’interprétation des données : | ||
| + | * Génomique des populations : détection de locus sous sélection, structuration génétique, inférences de paramètres démographiques. | ||
| + | * Génomique écologique : analyses d’association génotype-phénotype (ex : résistance à un pathogène) et génotype-environnement. | ||
| + | * Métagénomique (thématiques ciblées): détection de pathogènes dans des échantillons (alimentaires, archéologiques), quantification de la biodiversité dans des environnements cibles (systèmes aquatiques, sols,…), métagénomique des interactions (communautés, pollinisateurs). | ||
| + | * Paléogénomique : caractérisation des polymorphismes d’ADN anciens, estimation des phénomènes démographiques anciens (introgression, hybridation, …), détection de locus impliqués dans l’évolution adaptative d’un taxon. <br> | ||
| + | '''Références''' <br> | ||
| + | '''Mots Clefs''' : génomique des populations, génomique écologique, métagénomique, paléogénomique, Next-Generation Sequencing, Single Nucleotide Polymorphism (SNP), modélisation <br> | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Biologie des systèmes 1==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 2 parcours BBS''' <br> | ||
| + | '''ECTS : 6''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | Cet enseignement a pour but d'initier les étudiants à la biologie des systèmes dont l'objectif est de caractériser les composants élémentaires d'un système biologique (par exemple, une voie de régulation, une cellule, un écosystème etc.) pour mettre à jour les propriétés qui résultent de leurs interactions, ceci afin de mieux comprendre le comportement dynamique du système dans sa globalité. Dans cette UE, les problèmes de biologie des systèmes seront abordés via l’approche descendante ou ''top down'' (''versus'' l’approche ''bottom up''), qui s'appuie largement sur les résultats expérimentaux obtenus par les approches à haut débit. La compréhension d’un système va donc nécessiter d’intégrer les données obtenues à partir de différentes approches Omics, en fonction de la question abordée. Les concepts et méthodes nécessaires à l’analyse de ces différents types de données Omics seront détaillés et seront accompagnés par leur mise en pratique sur des cas concrets.<br> | ||
| + | '''Prérequis''' <br> | ||
| + | UE Traitement des données post-génomiques.<br> | ||
| + | '''Contenu''' <br> | ||
| + | Différentes problématiques scientifiques d’actualité en relation avec la biologie des systèmes seront abordées sous forme d'ateliers ce qui requerra une participation active des étudiants comme par exemple la préparation de l'atelier par la lecture et la présentation orale de publications scientifiques sur le sujet, la réalisation de projets personnels ou en groupe, etc. | ||
| + | Parmi les problématiques qui seront abordées, nous pouvons citer l’épigénomique, la métabolomique, l’étude des interactions ADN/protéines à l’échelle du génome, l’organisation spatiale de la chromatine dans la cellule, les approches « single cell ». Cette liste n’est pas exhaustive et pourra évoluer d’année en année en fonction de l’évolution de la discipline. Les concepts et méthodes nécessaires au traitement de ces nouveaux types de données Omics seront détaillés et seront accompagnés par leur mise en pratique pour aborder, par exemple, des questions en recherche biomédicale. | ||
| + | <br> | ||
| + | '''Références'''<br> | ||
| + | '''Mots Clefs''' : Biologie des systèmes ; intégration des données ; traitement des données ChIP-Seq ; Chromosome conformation capture ; épigénomique ; métabolomique ; single cell. | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Biologie des systèmes 2==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 2 parcours BBS''' <br> | ||
| + | '''ECTS : 6''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | Dans cette UE, les problèmes de biologie des systèmes seront abordés via l’approche ascendante ou ''bottom up'' (''versus'' l’approche ''top down'') dont le but est d’intégrer dans un modèle mathématique l’ensemble des données expérimentales obtenues sur un système d’étude pour pouvoir simuler le comportement dynamique de ce système au cours du temps mais aussi dans différentes conditions physiologiques et/ou environnementales. Les différents formalismes mathématiques disponibles pour modéliser un système biologique et le simuler seront décrits du point de vue théorique et mis en pratique sur des cas concrets de modélisation de processus biologiques. A l’issue de cette UE, les étudiant(e)s sont capables de construire un modèle, de choisir le bon formalisme mathématique en fonction des données expérimentales disponibles sur le processus (système) étudié et de simuler son comportement. <br> | ||
| + | '''Prérequis''' <br> | ||
| + | Notions de calcul matriciel; équations différentielles ordinaires<br> | ||
| + | '''Contenu''' <br> | ||
| + | Les enseignements aborderont la description des différentes méthodes d'analyse dynamique des réseaux biologiques et de prédiction de leur comportement. Les approches qualitatives ainsi que les approches quantitatives seront abordées. Dans le cas des approches qualitatives, la modélisation par équations différentielles linéaires par morceaux et les modèles logiques seront plus particulièrement développés. Pour les approches quantitatives, les modèles déterministes basés sur les équations différentielles ordinaires seront privilégiés. Les réseaux de Pétri, proposant un formalisme graphique et mathématique permettant de modéliser et simuler le comportement dynamique d'un système par une approche qualitative ou quantitative seront également décrits. | ||
| + | Le problème d’estimation des valeurs des paramètres du modèle quand elles ne sont pas connues expérimentalement sera abordé et quelques méthodes d’estimation de ces paramètres seront décrites. | ||
| + | Les TP aborderont des cas pratiques issus de divers domaines de la biologie comme : les réactions enzymatiques, la croissance de biomasse, l'analyse de processus périodiques et la modélisation de réseaux de régulation de gènes. <br> | ||
| + | '''Références''' <br> | ||
| + | An introduction to Systems Biology: Design principles of Biological Circuits (2007). Uri Alon. Chapman & Hall/CRC <br> | ||
| + | '''Mots Clefs''' : Biologie des systèmes ; systèmes biologiques dynamiques ; réseaux d'interactions ; modélisation ; simulation | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Ouverture : international, bioéthique, insertion professionnelle==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 2 parcours BBS et BGE''' <br> | ||
| + | '''ECTS : 3''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | Les objectifs de cette UE sont, d’une part, ouvrir l’esprit des futurs diplômés à des notions qui ne sont pas leur cœur disciplinaire et, d’autre part, développer les compétences transversales nécessaires à une insertion professionnelle réussie. <br> | ||
| + | Prérequis <br> | ||
| + | '''Contenu'''<br> | ||
| + | Les notions suivantes seront abordées à la fois sous forme d'enseignements traditionnels et de séminaires, y compris par des conférenciers internationaux : | ||
| + | * la bioéthique (en particulier nos droits et devoirs envers les organismes vivants, y compris les plus simples, le rapport du scientifique au public); | ||
| + | * intelligence artificielle : pouvoir et limites (notamment en matière d'éthique); | ||
| + | * les avancées les plus récentes dans le domaine des nanobiotechnologies : bases, concepts, applications.<br> | ||
| + | Enfin, en vue de leur insertion professionnelle, des tables rondes seront organisées avec d'anciens diplômés du Master, recrutés dans le monde académique ou dans le secteur privé : recherche d'emploi, stratégies de candidature, intégration, mise en réseau. Les étudiants seront interpellés à la fois par un questionnement personnel sur leurs objectifs professionnels et par des entretiens d'embauche fictifs.<br> | ||
| + | Références <br> | ||
| + | '''Mots Clefs''' : <br> | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Communication scientifique==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 2 parcours BBS et BGE''' <br> | ||
| + | '''ECTS : 3''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | L’objectif est d'apprendre et de mettre en œuvre la synthèse et la présentation de travaux scientifiques via différents supports de communication.<br> | ||
| + | '''Prérequis'''<br> | ||
| + | '''Contenu''' <br> | ||
| + | Elaboration de support de communication : diapositives, présentations interactives, poster. | ||
| + | Rédaction d'une synthèse de l'état de l'art sous forme d'une revue en anglais respectant un format de journal international. | ||
| + | En pratique, chaque étudiant se verra proposer une publication scientifique en relation avec les thématiques abordées dans les UE de biologie des systèmes 1 et 2 (parcours BBS), de méta-génomique écologique et évolutive (parcours BGE)et de phylogénomique. L'étudiant devra présenter cette publication à l'oral avec un support écrit en anglais. Le thème de cette publication servira de point de départ pour la synthèse et la rédaction de la mini-revue dont le format suivra celui demandé pour l'écriture d'une revue dans un journal international. <br> | ||
| + | '''Références''' <br> | ||
| + | <!-- competences=Produire une synthèse critique de la bibliographie relative au sujet étudié en ayant évalué la pertinence des informations collectées pour proposer de nouveaux développements et/ou aborder de nouvelles questions de recherche appliquée ou fondamentale.--> | ||
| + | '''Mots Clefs''' : analyse critique de publication ; rédaction scientifique ; recherche bibliographique ; présentation orale | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Stage académique==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 2 parcours BBS et BGE''' <br> | ||
| + | '''ECTS : 30''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | Le stage de 6 mois (de janvier à juin) en milieu académique permettra à l'étudiant d'apprendre à travailler en équipe, de gérer un projet de recherche et donc d'organiser son travail de manière à répondre aux échéances exigées par l'encadrant, de trouver des solutions pour résoudre des problèmes qui n'avaient pas été envisagés au départ. Il permettra aussi de mettre en oeuvre les compétences acquises lors de la formation académique pour résoudre des problématiques concrètes. | ||
| + | Le travail réalisé donnera lieu à un rapport écrit et à une soutenance orale. | ||
| + | <br> | ||
| + | '''Prérequis'''<br> | ||
| + | '''Contenu''' <br> | ||
| + | Travailler dans un environnement pluridisciplinaire (biologistes, bioinformaticiens, biostatisticiens, informaticiens) au sein d'une entreprise tout en faisant preuve d'autonomie et d'initiatives. | ||
| + | Défendre un projet et/ou des résultats en utilisant les moyens de communication adéquats lors de réunions d'équipe, conférences ou jurys.<br> | ||
| + | <!--'''Références''' <br> | ||
| + | competences=Produire une synthèse critique de la bibliographie relative au sujet étudié en ayant évalué la pertinence des informations collectées pour proposer de nouveaux développements et/ou aborder de nouvelles questions de recherche appliquée ou fondamentale.--> | ||
| + | '''Mots Clefs''' : immersion professionnelle. | ||
| + | </font> | ||
| + | |||
| + | <hr> | ||
| + | |||
| + | ====Stage professionnel==== | ||
| + | <font face = "Times New Roman" size = "3"> | ||
| + | '''Niveau : Master 2 parcours BBS et BGE''' <br> | ||
| + | '''ECTS : 30''' <br> | ||
| + | '''Objectifs''' <br> | ||
| + | Le stage professionnel de 6 mois (de janvier à juin) en entreprise ou milieu académique permettra à l'étudiant d'apprendre à travailler en équipe, de gérer un projet de développement logiciel lié à une question biologique et donc d'organiser son travail de manière à répondre aux échéances exigées par l'encadrant, de trouver des solutions pour résoudre des problèmes qui n'avaient pas été envisagés au départ. Il permettra aussi de mettre en oeuvre les compétences acquises lors de la formation académique pour résoudre des problématiques concrètes. | ||
| + | Le travail réalisé donnera lieu à un rapport écrit et à une soutenance orale. | ||
| + | <br> | ||
| + | '''Prérequis'''<br> | ||
| + | '''Contenu''' <br> | ||
| + | Travailler dans un environnement pluridisciplinaire (biologistes, bioinformaticiens, biostatisticiens, informaticiens) au sein d'une entreprise tout en faisant preuve d'autonomie et d'initiatives. | ||
| + | Défendre un projet et/ou des résultats en utilisant les moyens de communication adéquats lors de réunions d'équipe, conférences ou jurys. | ||
| + | . <br> | ||
| + | <!-- '''Références''' <br> | ||
| + | competences=Produire une synthèse critique de la bibliographie relative au sujet étudié en ayant évalué la pertinence des informations collectées pour proposer de nouveaux développements et/ou aborder de nouvelles questions de recherche appliquée ou fondamentale.--> | ||
| + | '''Mots Clefs''' : immersion professionnelle. | ||
| + | </font> | ||
| + | <!-- == Master 1 == | ||
Le premier semestre permet aux étudiants d'acquérir les fondements disciplinaires de la formation (informatique, mathématiques et bioinformatique). | Le premier semestre permet aux étudiants d'acquérir les fondements disciplinaires de la formation (informatique, mathématiques et bioinformatique). | ||
| Ligne 86 : | Ligne 957 : | ||
| − | [[Image:Schema de la mention Bioinformatique.png|thumb|100px|Schéma de la formation]] | + | [[Image:Schema de la mention Bioinformatique.png|thumb|100px|Schéma de la formation]] --> |
= Historique de la formation = | = Historique de la formation = | ||
| Ligne 92 : | Ligne 963 : | ||
* 2005 : Réforme LMD, le DESS devient un Master 2 Professionnel | * 2005 : Réforme LMD, le DESS devient un Master 2 Professionnel | ||
* 2011 : Evolution du Master sur deux années de formation M1-M2I au sein de la mention Microbiologie-AgroBiosciences-Biologie des Systèmes (MABS) | * 2011 : Evolution du Master sur deux années de formation M1-M2I au sein de la mention Microbiologie-AgroBiosciences-Biologie des Systèmes (MABS) | ||
| − | * 2016 : Mention Bioinformatique | + | * 2016 : Mention Bioinformatique parcours Bioinformatique et Biologie des Systèmes |
| + | * 2022 : Création d'un second parcours Bioinformatique et Génétique Environnementale au sein de la mention Bioinformatique | ||
| + | |||
Jusqu’en 2015 la mention Bioinformatique était enregistrée sous la dénomination mention Microbiologie-AgroBiosciences-Biologie des Systèmes (MABS) et était constitué par le parcours Bioinformatique et Biologie des Systèmes en première année de master et par la spécialité Bioinformatique et Biologie des Systèmes en deuxième année de master. | Jusqu’en 2015 la mention Bioinformatique était enregistrée sous la dénomination mention Microbiologie-AgroBiosciences-Biologie des Systèmes (MABS) et était constitué par le parcours Bioinformatique et Biologie des Systèmes en première année de master et par la spécialité Bioinformatique et Biologie des Systèmes en deuxième année de master. | ||
Version actuelle en date du 24 juin 2022 à 10:44
Sommaire
- 1 Schéma général des études
- 1.1 Syllabus
- 1.2 Le parcours Bioinformatique et Biologie des Systèmes
- 1.3 Le parcours Bioinformatique et Génomique Environnementale
- 1.4 Description des Unités d'Enseignement
- 1.4.1 UE Master 1
- 1.4.1.1 Algorithmique et complexité
- 1.4.1.2 Programmation en bioinformatique
- 1.4.1.3 Harmonisation des connaissances en biologie
- 1.4.1.4 Mathématiques pour la biologie
- 1.4.1.5 Traitement des données biologiques
- 1.4.1.6 Bioinformatique des séquences
- 1.4.1.7 Génétique évolutive et quantitative
- 1.4.1.8 Introduction aux bases de données
- 1.4.1.9 Bioinformatique pour la Génomique
- 1.4.1.10 Traitement de graphes et réseaux biologiques
- 1.4.1.11 Bioanalyse
- 1.4.1.12 Analyse de données et modélisation en écologie
- 1.4.1.13 Ecologie évolutive
- 1.4.1.14 Ecologie des communautés et fonctionnement des écosystèmes
- 1.4.1.15 Programmation avancée et génie logiciel
- 1.4.1.16 Génomique Environnementale
- 1.4.1.17 Fouille de données
- 1.4.1.18 Traitement de données post-génomiques
- 1.4.1.19 Evolution moléculaire
- 1.4.1.20 Projet tutoré en bioinformatique
- 1.4.1.21 Introduction Modélisation Moléculaire
- 1.4.1.22 Analyse de données multivariées
- 1.4.1.23 Biostatistiques : Utilisation avancée du modèle linéaire
- 1.4.1.24 Stage (facultatif)
- 1.4.2 UE Master 2
- 1.4.2.1 Bases de données avancées
- 1.4.2.2 Introduction Apprentissage automatique
- 1.4.2.3 Gestion de données non structurées et applications post-génomiques
- 1.4.2.4 Phylogénomique
- 1.4.2.5 Métagénomique écologique et évolutive
- 1.4.2.6 Biologie des systèmes 1
- 1.4.2.7 Biologie des systèmes 2
- 1.4.2.8 Ouverture : international, bioéthique, insertion professionnelle
- 1.4.2.9 Communication scientifique
- 1.4.2.10 Stage académique
- 1.4.2.11 Stage professionnel
- 1.4.1 UE Master 1
- 2 Historique de la formation
Schéma général des études
Le master de Bioinformatique se transforme à la rentrée 2022 et s'enrichit d'un second parcours, le parcours de Bioinformatique et Génomique Environementale en complément de celui déjà existant, le parcours de Bioinformatique et Biologie des Systèmes.
La plaquette du master fournit un résumé descriptif de cette nouvelle offre de formation
Cette section est donc en cours de construction et s'enrichira au cours du temps.
Le schéma ci-dessous présente l’organisation en UE de la mention par année et parcours :
- En vert les UE spécifiques au parcours « Bioinformatique et Génomique Environnementale » (BGE)
- En mauve les UE spécifiques au parcours « Bioinformatique et Biologie des Systèmes » (BBS)
- En gris les UE mutualisées entre les deux parcours
Syllabus
Les syllabus ne sont pas encore disponibles sur le site de l'Université Paul Sabatier. En attendant leurs mises en ligne, merci de vous référer à la section Description des Unités d'Enseignements ci-dessous.
Le parcours Bioinformatique et Biologie des Systèmes
Objectifs de formation
Le parcours BBS permettra aux étudiants d’acquérir (1) les connaissances en programmation et gestion des données pour accompagner des projets en biologie, (2) les compétences en traitements mathématiques de grands jeux de données pour en extraire les informations pertinentes, (3) les démarches pour dégager, à partir de différentes sources de données hétérogènes, les relations entre objets dans le but d'inférer des réseaux biologiques, (4) les méthodes de modélisation dynamique des réseaux biologiques pour analyser in silico leur comportement.
Contenu des enseignements
Ce parcours proposera une solide formation disciplinaire en Bioinformatique et en Biologie des Systèmes. Il mettra l'accent sur le traitement et l’intégration des différents types de données Omics, et sur l’inférence et la modélisation dynamique des réseaux de gènes afin de mieux comprendre le comportement dynamique du système biologique dans sa globalité.
Les principaux axes d’enseignement seront :
- en Informatique : Algorithmique et complexité, Programmation en Bioinformatique et Programmation avancée et Génie Logiciel, Introduction en bases de données et bases de données avancées,Gestion de données non structurées et les applications post-génomiques, Introduction à l'apprentissage automatique
- en Mathématiques : Mathématiques pour la Biologie, Traitement des données Biologiques, Analyses des données multivariées, Biostatistiques : utilisation avancée du modèle linéaire
- en Bioinformatique : Bioinformatique des séquences, Bioinformatique pour la Génomique, Traitement des graphes et réseaux, Traitement des données post-génomiques, Fouille de données
- en Evolution : évolution moléculaire, phylogénomique, Génétique évolutive et quantitative
- en Biologie des Systèmes
A la place de l'UE d'algorithmique et complexité, une UE Harmonisation des connaissances en Biologie sera proposée aux étudiant(e)s venant d'une licence d'Informatique.
La pédagogie fait appel le plus largement possible au travail en autonomie et/ou en groupe et s'appuie sur l'analyse des processus d'acquisition des connaissances scientifiques, des ateliers pratiques et la réalisation de projets. Le second semestre est consacré à un stage long (6 à 5 mois) dans des laboratoires de recherche ou en entreprise.
Le parcours Bioinformatique et Génomique Environnementale
Ce parcours, mutualisé entre le master de Bioinformatique et le master Biologie, Écologie et Évolution (BEE), est donc également proposé par le master BEE.
Objectifs de formation
Le parcours BGE permettra aux étudiants d’acquérir des bases conceptuelles et pratiques solides qui leur permettront de (i) traiter des données génomiques issues de prélèvements environnementaux, (ii) décrire la diversité taxonomique et fonctionnelle de ces prélèvements, (iii) modéliser les interactions entre les différentes composantes des systèmes biologiques décrits, ainsi qu’inférer leur évolution et prédire leur capacité d’adaptation, à l’aide de méthodes statistiques et d’apprentissage automatique.
Contenu des enseignements
Ce parcours proposera une solide formation disciplinaire en Bioinformatique, Biodiversité, Ecologie et Evolution, pour permettre de générer des connaissances sur le fonctionnement et l’évolution des organismes et écosystèmes, par l’analyse de l’information génétique ou génomique issue du séquençage non ciblé, dans un environnement donné.
Ainsi les principaux axes d’enseignement porteront sur :
- La génomique environnementale (ADN environnemental, ADN ancien, métabarcoding, métagénomique, génomique des populations, génomique écologique, génomique comparative)
- L’algorithmique et la programmation en bioinformatique, le traitement des données génomiques et post-génomiques, la phylogénomique, le traitement des graphes et réseaux.
- Les concepts clés en écologie évolutive, écologie microbienne et écologie des communautés et sur le fonctionnement des écosystèmes.
- Les traitements et modélisation statistiques des données en écologie et les modélisations par apprentissage automatique.
La pédagogie fait appel le plus largement possible au travail en autonomie et/ou en groupe et s'appuie sur l'analyse des processus d'acquisition des connaissances scientifiques, des ateliers pratiques et la réalisation de projets. Le second semestre est consacré à un stage long (6 à 5 mois) dans des laboratoires de recherche ou en entreprise.
Description des Unités d'Enseignement
Le parcours et l'année sont indiquées pour chaque UE
UE Master 1
Algorithmique et complexité
Niveau : Master 1 parcours BBS et BGE
ECTS : 3
Objectifs
Cette UE est pour les étudiants provenant de biologie. Les autres étudiants suivront l'UE d'Harmonisation des connaissances en biologie.
Décomposer un programme en éléments de plus petite taille.
Analyser le comportement de programmes simples fondés sur les variables, expressions, affectations, E/S, structure de contrôle, fonctions...
Créer des algorithmes résolvant des problèmes simples : transcription ADN en ARN, traduction en protéines.
Implémenter des algorithmes, les tester et les déboguer.
Comprendre les notions et les techniques qui permettent d’analyser la complexité d’algorithmes.
Initiation aux systèmes Unix : gestion des fichiers, commandes de base.
Prérequis
Mathématiques élémentaires
Contenu
Algorithmique
- Syntaxe élémentaire, notion de variables, constantes...
- Expressions et affectations
- Entrées-sorties simples
- Structures de contrôle
- Types de données (entiers, réels, chaîne de caractères, tableaux, listes, dictionnaires)
- Fonctions
Concepts de la programmation
- Concept d'algorithme
- Types d'erreur
- Compréhension des programmes
- Algorithmes simples (moyenne, min, max d’une liste, manipulation de séquences ADN, transcription ARN, traduction protéines)
- Stratégies de résolution de problèmes
Complexité
Introduction des notions et techniques qui permettent d’analyser la complexité d’algorithmes.
Complexité de boucles, pour ou tantque, dans le cas le pire et le cas moyen.
Systèmes Unix
- introduction aux système de gestion de fichiers
- commandes SHELL de base, notions de filtrages et d'entrées sorties
Références
Algorithmes - Notions de base. Thomas H. Cormen. Dunod, 2013.
Unix, Initiation et utilisation : Colin, Armspach, Ostré-Waerzeggers, Dunod, 2004
Programmation en bioinformatique
Niveau : Master 1 parcours BBS et BGE
ECTS : 3
Objectifs
Apprentissage de la programmation structurée avec des langages impératifs. Savoir utiliser les structures de contrôle (tests, boucles), les types élémentaires et les structures de données classiques (listes, dictionnaires, etc). Savoir structurer son code en fonctions et transformer les scripts en modules réutilisables. Mise en œuvre en utilisant un langage impératif (python). Utilisation de modules/paquets/bibliothèques de programmation dédiés à la bioinformatique, notamment pour la manipulation de séquences (nucléiques et protéiques) en utilisant les formats standards en bioinformatique (FASTA, GenBank, etc), ainsi que l’exploitation de diverses sources et formats de données. Introduction à la programmation objet. Savoir utiliser une interface de programmation.
Prérequis
UE Algorithmique et complexité
Contenu
Notions fondamentales
- Expressions : types de données élémentaires, affectation, opérateurs arithmétiques
- Conditions : structures conditionnelles / expressions booléennes
- Itérations : structures itératives, types listes et chaînes de caractères
- Fonctions définies par le programmeur
- Gestion des entrées et sorties : interactif, lecture et écriture de fichiers, formats compressés
Notions complémentaires
- Types structurés : listes, dictionnaires
- Expressions régulières (module re)
- Passage d’options et d’arguments à un script (sys, argparse)
- Modules : importation et création
- Modules spécifiques : biopython (bioinformatique), matplotlib (graphique), numpy (algèbre), sklearn (analyse de données, apprentissage automatique)
Applications en bioinformatique
- Algorithmes de tri, de comptage, de recherche et de comparaison de séquences nucléiques et protéiques
- Transcription et traduction de séquences
- Lecture/écriture de fichiers FASTA et GenBank, extraction d’informations dans ce type de fichiers
Références
Apprendre à programmer avec Python 3, Gérard Swinnen, Eyrolles
Programmation en Python pour les sciences de la vie, Patrick Fuchs, Pierre Poulain, DUNOD
Mots clefs :
Algorithmique ; structures ; instructions conditionnelles ; programmation impérative ; Biopython ; séquences génomiques nucléiques et protéiques ; annotations structurées ;
Harmonisation des connaissances en biologie
Niveau : Master 1 parcours BBS
ECTS : 3
Niveau : Master 1 parcours BBS
Objectifs
Cette UE est pour les étudiants ne provenant pas de biologie. Les étudiants venant de biologie suivront l'UE d'Algorithmique et complexité. L’objectif est de fournir un enseignement de mise à niveau en Biologie Cellulaire, Biologie Moléculaire et Génétique à des étudiants venant de licences en Physique ou en Informatique.
Prérequis
Contenu
Biologie Cellulaire : Introduction générale à la biologie cellulaire. Notion de cellule procaryote et eucaryote. Organisation de la cellule (compartimentation et dynamique intracellulaire). Expression génique et régulation. Mouvements cellulaires. Prolifération, différenciation et mort cellulaire. Enseignement intéractif privilégiant un travail collectif.
Analyse Génétique : Relation gène-fonction. Notions de mutation et d’allèle. Lois de transmission de caractères héréditaires chez les eucaryotes (i.e. : monogénique et digénique). Notions d’indépendance et de liaison génique. Transmission génétique chez les procaryotes.
Biologie Moléculaire : Structure du matériel génétique, notions de gène et de génome. Présentation des grands processus moléculaires de la cellule (i.e. : réplication, transcription, traduction).
Références
Biologie (Campbell et Reece - De Boeck)
competences=Niveau de base (licence) en Biologie Moléculaire, Biologie Cellulaire et Génétique -->
Mots Clefs :
Biologie cellulaire ; Biologie Moléculaire ; Génétique mendélienne et moléculaire
Mathématiques pour la biologie
Niveau : Master 1 parcours BBS
ECTS : 3
Objectifs
Initiation théorique aux bases d'algèbre linéaire, d'analyse et de probabilités. Représentation matricielle. Modélisation de problèmes dynamiques : résolution d'équations différentielles. Présentation des algorithmes numériques pour résoudre ces problèmes. Probabilités. Mise en application dans le cadre d’un projet applicatif en biologie des notions théoriques et algorithmes vus en cours.
Prérequis
Contenu
Algèbre Linéaire
- Opérations élémentaires sur les matrices
- Déterminant
- Diagonalisation : recherche de vecteurs/valeurs propres.
- résolutions de systèmes linéaires,
Analyse
- intégration,
- équations différentielles,
- optimisation,
- Droite de régression : problème des moindres carrés ordinaires
Probabilités
- Probabilités conditionnelles, règles de Bayes
- Variables aléatoires discrètes et continues : lois de probabilités, espérances, variances, covariance, corrélation
- modèle linéaire
Références
Mathématiques pour les Sciences de la vie et de la Terre – C. David, S. Mustapha, F. Viens, N. Capron, edition Dunod
Mots-clés : systèmes linéaires ; vecteurs/valeurs propres ; équations différentielles ; probabilités
Traitement des données biologiques
Niveau : Master 1 parcours BBS
ECTS : 3
Objectifs
Acquisition des notions fondamentales de l’analyse statistique appliquée aux données biologiques, et maîtrise de l’utilisation du logiciel R/Rstudio. Les étudiants doivent pouvoir (i) décrire des données biologiques associées à un plan expérimental, (ii) tester des hypothèses biologiques liées aux données par des méthodes statistiques adaptées, et (iii) interpréter biologiquement les résultats de ces traitements.
Prérequis
Connaissances de base en mathématiques. Notion d'expression génique.
Contenu
- Statistiques descriptives (une ou plusieurs variables qualitatives et/ou quantitatives, représentations graphiques, introduction aux analyses multivariées).
- Statistiques inférentielles (loi de probabilité, distribution d’échantillonnage, estimation ponctuelle, estimation par intervalle de confiance, tests paramétriques et non paramétriques, tests multiples).
- Introduction au traitement des données post-génomiques par l’analyse de données multivariées telles que des données de transcriptome : différence d’expression des gènes, recherche de co-régulations géniques par analyses multivariées et clustering, caractérisation d’ensembles de gènes.
Références
The R Book (Mickael J Crawley)
Mots-clés : statistiques descriptives ; probabilités ; échantillonnage ; test statistique ; analyses multivariées ; partitionnement
Bioinformatique des séquences
Niveau : Master 1 parcours BBS
ECTS : 3
Objectifs
Comprendre les concepts et algorithmes sous-jacents aux principaux outils de comparaison de séquences biologiques afin d’être capable de choisir la méthode la plus pertinente pour répondre à une problématique.
Prérequis
Connaissance de biologie moléculaire et de bases en bioanalyse (contenu des banques, interrogation, recherche d'homologues).
Contenu
Les cours viseront à montrer la différence entre un alignement exact (Needleman et Smith et Waterman) ou approché via des heuristiques (type BLAST) ; l'intérêt de la méthode de programmation dynamique pour la comparaison de séquences ; comprendre les différentes approches en alignement multiple de séquences : méthodes locales et globales, progressives versus itératives ; connaître les méthodes de caractérisation (signature, profile) et la recherche de motifs communs entre plusieurs séquences.
La mise en pratique de ces différentes méthodes sera faite lors de séances de travaux dirigés et pratiques, en insistant sur leurs avantages ou leurs limites.
Références
Bio-informatique. Principes d'utilisation des outils. 2010. D. Tagu, J.L. Risler, coord. Ed Quae
Bioinformatique - Cours et applications. 2ème édition. 2015. G. Deléage et M. Gouy. Dunod
Mots clefs : Algorithmes pour la comparaison de séquences ; heuristique ; profils
Génétique évolutive et quantitative
Niveau : Master 1 parcours BBS
ECTS : 3
Objectifs
Acquisition des concepts fondamentaux de la génétique évolutive (génétique des populations) et de la génétique quantitative (architecture génétique des caractères quantitatifs, cartographie de QTL, association génotype/phénotype). Analyse de données génétiques et phénotypiques.
Prérequis
Notion de génétique Mendélienne (ségrégation des caractères) et de polymorphisme moléculaire.
Contenu
- Génétique des populations : mesures du polymorphisme génétique, modèle de Hardy-Weinberg, systèmes de reproduction (autogamie, consanguinité, ...), forces évolutives (mutation, sélection, dérive génétique, migration).
- Génétique quantitative : modèle polygénique des caractères quantitatifs, modèle qualitatif, notion d’héritabilité, notion de ressemblance / apparentement génétique, principes de la cartographie de Quantitative Trait Loci (QTL) et de la génétique d’association.
- Ce contenu sera traité au travers d’exemples issus du monde végétal et de l’agronomie.
Références
Précis de génétique des populations (Jean-Pierre Henry, Pierre-Henry Gouyon, ed Dunond)
Introduction to quantitative genetics (Falconer & Mackay) Handbook of statistical genetics (Balding, Bishop, Cannings)
Mots Clefs : génétique ; population ; évolution ; caractère quantitatif ; cartographie génétique
Introduction aux bases de données
Niveau : Master 1 parcours BBS et Master 2 parcours BGE
ECTS : 3
Objectifs
L'objectif de ce cours est d'apprendre aux étudiants à concevoir une base de données et de l’interroger en algèbre relationnelle ainsi qu'en langage déclaratif tel que le langage SQL.
Prérequis
Contenu
- Définition, objectifs des bases de données et fonctions des SGBD
- Modèles de données
- Modèle conceptuel : modèle Entité/Association E/A
- Modèle logique : modèle relationnel
- Algèbre relationnelle
- Langages de définition et de manipulation des bases de données relationnelles
- LDD : Langage de Définition de Données
- LMD: Langage de Manipulation de Données
Références
Gardarin G., "Bases de données", Edition Eyrolles, 2003 (ISBN 2-212-11281-5).
Mots Clefs : bases de données relationnelles ; modélisation ; algèbre relationnelle ; LDD ; LMD ; language SQL
Bioinformatique pour la Génomique
Niveau : Master 1 parcours BBS et Master 2 parcours BGE
ECTS : 3
Objectifs
Cet enseignement permettra aux étudiants d'acquérir les approches d'analyses des données de génomes, plus particulièrement l'annotation des séquences génomiques et la génomique comparative. Les concepts et les questions biologiques sous-jacents à ces approches seront abordés et seront suivis de leur mise en pratique sur des cas concrets.
Prérequis
Contenu
Les cours aborderont :
- La description de la conception d'un prédicteur de gène et des différentes méthodes qui doivent être mise en œuvre pour effectuer les mesures nécessaires sur la séquence génomique analysée (type contenu, signal, similarité) et comment ces différentes informations sont intégrées dans un modèle de structure de gène qui sera ensuite implémenté dans une solution logicielle permettant de réaliser la prédiction de la structure optimale. Parmi ces approches, les modèles de Markov cachés (HMM) permettant de réaliser des modèles probabilistes d’une suite de problèmes linéaires labellisés seront plus particulièrement développés.
- la description des concepts et des hypothèses fonctionnelles qui sous-tendent les approches de génomique comparative.
Les séances de TP auront lieu sur ordinateur de manière à mettre en pratique les approches et démarches théoriques décrites en cours.
Références
Biological sequences analysis: Probabilistic Models of Proteins and Nucleic Acids (1998) R. Durbin, S. Eddy, A. Krogh, G. Mitchison. Cambridge University Press.
Mots Clefs : Annotation de génomes ; Modèle de Markov caché ; génomique comparative ; alignement de génomes ; synténie
Traitement de graphes et réseaux biologiques
Niveau : Master 1 parcours BBS et Master 2 parcours BGE
ECTS : 3
Objectifs
Dans ce module, les étudiants aborderont les concepts et les algorithmes de base en théorie des graphes. Quelques problèmes classiques de biologie seront revisités à la lumière de ces concepts. Les réseaux d’interactions moléculaires (régulation transcriptionnelle, réseaux métaboliques, réseaux d’interactions protéiques), les ontologies liées aux représentations des concepts biologiques, et les problèmes de graphes associés constitueront des applications privilégiées.
Prérequis
UE de programmation en bioinformatique
Contenu
- Concepts et définitions
- Graphes et réseaux rencontrés en biologie et bioinformatique
- Représentations informatiques et manipulations
- Formats de stockage
- Logiciels de visualisation, d'édition et d'analyse
- Librairies et boites à outils
- Algorithme de parcours et de dessin
- Arbres couvrants
- Détection de motifs
- Partitionnement et détection de communautés
Références
Introduction to Algorithms, Corsen, Leiserson and Rivest, MIT Press and McGraw-Hill
Mots-clés : parcours ; plus courts chemins ; marches aléatoires ; partitionnement de graphes ; détection de communautés ; modularité ; réseau d'interaction ; réseau de régulation ; réseau métabolique
Bioanalyse
Niveau : Master 1 parcours BGE
ECTS : 3
Objectifs
Cet enseignement a pour but de former les étudiants aux approches bio-informatiques utilisées dans le cadre de l'analyse de séquences biologiques. Les concepts sous-jacents à ces approches seront décrits et seront suivis de leur mise en pratique.
Prérequis
Utilisation d'un navigateur web, notion de Biologie Moléculaire (ADN, ARN, protéines…)
Contenu
Les cours viseront en une présentation : des différentes banques de données et des systèmes d'interrogation ; des méthodes de recherche par similarité dans ces banques (Blast, Psi-Blast); des méthodes utilisées pour la comparaison de deux séquences (matrice de points, alignement global et local) ainsi que celles développées pour les alignements multiples; des matrices de substitution (PAM, BLOSUM etc..) ; des approches dédiées à la recherche de motifs, signatures et profils.
Les séances sur ordinateur illustreront les démarches et approches de bioanalyse décrites en cours, en utilisant des logiciels dédiés et des données biologiques disponibles dans les bases de données.
Références
Cours et applications (2015). Gilbert Deléage et Manolo Gouy. 2ème édition. DUNOD
Mots Clefs :
Banque, BLAST, alignements, domaines et motifs.
Analyse de données et modélisation en écologie
Niveau : Master 1 parcours BBS et BGE
ECTS : 6
Objectifs
Maîtriser les méthodes d'analyse de données (outils statistiques et de modélisation) les plus communément utilisées pour l'analyse et la compréhension des phénomènes biologiques et écologiques. Faire le lien entre le questionnement biologique et les méthodes d'analyse à mettre en œuvre afin de maîtriser l'ensemble de la démarche expérimentale, depuis la formulation des hypothèses biologiques jusqu'à l'interprétation des résultats, en passant bien évidemment par l'analyse des données. Acquérir une bonne autonomie dans les analyses de données permettant de répondre à des problématiques écologiques simples mais variées.
Prérequis
Avoir déjà suivi un enseignement sur les probabilités, les statistiques descriptives et les tests d'hypothèses. Avoir des connaissances de base du logiciel R.
Contenu
Les concepts et méthodes d'analyse abordés concerneront les lois de probabilité, les tests d'hypothèses (paramétriques et non paramétriques), le modèle linéaire (plans expérimentaux, analyse de variance, régression simple et multiple), le modèle linéaire généralisé, la classification et l'analyse en composantes principales. Les aspects théoriques indispensables à la compréhension et à la bonne utilisation de ces méthodes seront traités au cours des CM. Les TP, qui auront lieu en salle informatique, permettront aux étudiants de mettre en pratique ces méthodes afin de répondre à des problématiques biologiques concrètes. Pour cela, ils seront familiarisés à un outil de traitement de données gratuit (le logiciel R). Une place importante sera donnée à l'interprétation écologique des résultats statistiques. Enfin, un projet réalisé en fin d'UE par binôme permettra aux étudiants d'être confrontés à un problème biologique concret. Ils devront ainsi identifier les méthodes d'analyse adaptées à la question biologique posée et acquérir les bons « réflexes » face aux difficultés de l'analyse de données en situation réelle.
Références
Bertrand F. & Maumy-Bertrand M. 2014. Initiation à la statistique avec R. Cours et exercices corrigés. Dunod.
Scherrer B. 2008. Biostatistique volume 1. Editeur Gaëtan Morin.
Crawley M.J. 2012. The R Book. Wiley.
Mots Clefs : Analyses univariées et bivariées, modélisation, tests d'hypothèses, programmation, simulation.
Ecologie évolutive
Niveau : Master 1 parcours BGE
ECTS : 6
Objectifs
- Caractériser les variations (d'effectifs, de génotypes, de phénotypes et d'histoires de vie) dans les populations naturelles,
- Comprendre les mécanismes qui génèrent, maintiennent et modifient ces variations,
- Maîtriser les outils de description et d'analyse de ces variations
Prérequis
Maîtrise des concepts basiques en écologie, biologie évolutive, génétique, dynamique des populations, statistique inférentielle, mathématiques bac S.
Contenu
- Sélection naturelle (concepts et exemples)
- Dynamique des populations
- Génétique des populations (forces évolutives, structuration, régimes de reproduction, déséquilibre de liaison, coalescence)
- Dynamique adaptative (introduction)
- Génétique quantitative (héritabilité, équation du sélectionneur)
- Evolution des histoires de vie (mortalité, fécondité, itéroparité/semelparité, sénescence, âge à maturité, longévité…)
- Allocation au sexe de la descendance
- Dynamique et évolution des interactions (co-évolution, co-adaptation)
- Evolution expérimentale (expériences de terrain et en laboratoire, sélection artificielle)
Références
Thomas, F. et al. 2010. Biologie évolutive. Ed De Boeck
Mayhew, P. J. 2006. Discovering Evolutionary Ecology: Bringing Together Ecology and Evolution. Oxford Univ. Press.
Ricklefs, R. E., et Miller, G. L. 2005. Écologie. Ed De Boeck.
Mots Clefs : Sélection, dynamique et génétique des pop., génétique quantit., dynamique adaptative, interactions, évolution expérimentale, histoire de vie, allocation au sexe.
Ecologie des communautés et fonctionnement des écosystèmes
Niveau : Master 1 parcours BGE
ECTS : 6
Objectifs
Les cours magistraux et les travaux dirigés apportent les bases conceptuelles et empiriques indispensables à la compréhension des règles d'assemblages des communautés et des forces régissant le fonctionnement des écosystèmes. L'écologie est présentée comme une discipline intégrative profondément enracinée dans les sciences naturelles, mais dont les bases théoriques dérivent de principes généraux issus de la biologie, de la physique et de la chimie. Les enseignements apportent aussi un éclairage sur l'écologie en tant que science appliquée au service de l'humanité.
Prérequis
Connaissances académiques en biologie des organismes et des populations. Compréhension de la démarche scientifique et des méthodes d'analyse de données.
Contenu
Cours :
- Les multiples facettes de la biodiversité et leurs relations
- Le concept de niche écologique : émergence, évolution, et applications
- Règles d'assemblage et de dé-sassemblage des communautés
- Dynamique des réseaux d'interactions
- Evolution et stabilité des écosystèmes
- Cycle de la matière organique; cycles des éléments et stoechiomètrie écologique
- Rôle de la biodiversité dans le fonctionnement des écosystèmes et les services écologiques
- Influence de la structure de l'habitat, des facteurs physico-chimiques et des communautés dans le fonctionnement des sols et des systèmes aquatiques
- Ecologie appliquée : restauration et ingénierie écologique
- L'écologie à l'échelle du paysage et des méta-écosystèmes
Travaux dirigés :
- Outils numériques pour l'étude de la biodiversité et la modélisation des écosystèmes
- Utilisation des biomarqueurs et des bioindicateurs en écologie
Références
Weathers K.C. et al. (2013) Fundamentals of Ecosystem Science. Academic Press
Morin P.J. (2011) Community Ecology, Wiley-Blackwell
Naeem S. et al. (2009) Biodiversity, Ecosystem Functioning, & Human Wellbeing: Oxford University Press
Mots Clefs : biodiversité, écosystème, autoécologie, synécologie, traits fonctionnels, cycle de la matière organique et des nutriments, ingénierie écologique.
Programmation avancée et génie logiciel
Niveau : Master 1 parcours BBS
ECTS : 6
Objectifs
- Maitriser la modélisation objets d’un programme informatique
- Connaitre les différents concepts de la programmation orientée objet
- Savoir développer une application en utilisant une approche orientée objet
- Maitriser l’utilisation d’API dans le développement d’un programme informatique
- Connaitre les principaux éléments permettant de concevoir et développer une interface graphique
Prérequis
- Bonne connaissance des base de l’algorithmique
- Première expérience avec un langage de programmation|
Contenu
Partie génie logiciel : Modélisation avec UML
- Introduction sur l'intérêt de la modélisation
- Présentation des cas d'utilisation et des principaux diagrammes (diagramme de classe, de séquence, d’état transition)
Partie programmation orientée objet - Langage JAVA
- Introduction du langage JAVA
- Présentation des différents outils de compilation, éxecution, documentation, etc. et des principaux environnements de programmation (Eclipse, NetBeans)
- Notion de classe et objet
- Principales caractéristiques d'une classe (notion d'attributs, constructeurs, méthodes, visibilité)
- Instanciation des classes,
- Utilisation de package
- Lien avec la modélisation UML
- Notion avancée de programmation orientée objet : héritage, interface, classe abstraite
- Gestion des exceptions
- Présentation d'API
- Différentes structures de données
- Gestion des flux de données : utilisation de fichiers, lecture et écriture dans des flux, sérialisation, parseur XML
- Introduction aux interfaces graphiques et à la programmation événementielle
Références
Mots Clefs : Modélisation objet ; UML ; Programmation orientée objet ; JAVA ; Classes ; Objets ; Héritage
Génomique Environnementale
Niveau : Master 1 parcours BGE
ECTS : 6
Objectifs
- Mobiliser les concepts fondamentaux relatifs à la génomique environnementale, les objets qu’elle permet d’étudier (communauté complexe, génome d’organismes non modèles, niveau de variabilité, ADN ancien, détection d’espèces rares), les conditions de mise en oeuvre (échantillonnage, choix des marqueurs et des techniques de séquençage), les outils bioinformatiques nécéssaires (différents pipelines), les limitations et précautions (variabilité intra et interspécifique, contaminations, seuils…)
- Identifier, choisir une combinaison de méthodes et d’outils analytiques relatifs aux NGS pour répondre à des questions d’écologie et d’évolution.
- Interpréter des résultats issus de l’analyse de données de génomique environnementale - qu’ils soient originaires d’un article ou des analyses menées par les étudiants.
- Exploiter des logiciels d’acquisition et d’analyse de données NGS avec un esprit critique.
Prérequis
Contenu
Les cours magistraux permettront d’introduire la génomique environnementale, ses questions, ses objets, ses technologies, puis d’explorer trois grands thèmes autour des usages de la génomique environnementale pour étudier (1) l’ADN ancien ou dégradé, (2) l’écologie microbienne et les interactions, et enfin (3) la comparaison des génomes, tant d’eucaryotes que de procaryotes.
Les TD seront dédiés à la présentation de cas d’étude sous forme d’article, pour pouvoir décrypter l’ensemble des étapes d’un projet de génomique environnementale, avec un oeil critique. Un des TD sera aussi dédié à une visite virtuelle ou non d’une plateforme de séquençage. L’application de ces techniques dans le domaine de l’écologie de la conservation sera aussi abordée dans le cadre des TD.
Enfin les TP aborderont deux des trois grands thèmes présentés, et permettront d’appliquer des outils bioinformatiques, et décrypter les lignes de commandes, et d’aboutir à des résultats que les étudiants devront interpréter.
Références
Mots Clefs
Fouille de données
Niveau : Master 1 parcours BBS et BGE
ECTS : 3
Objectifs
Cette UE a pour but d’initier les étudiants aux techniques modernes de fouilles de données permettant de prédire, par exemple, si celui qui achète du pain et du beurre va acheter de la confiture, s’il est raisonnable pour une banque d'attribuer une carte de crédit, si la protéase PfSUB1 joue un rôle dans la division de Plasmodium falciparum (parasite responsable de la malaria), ou encore de diagnostiquer un sous-type de cancer du sein à partir de données transcriptomique de la patiente. En général, plusieurs méthodes peuvent être utilisées, plus ou moins performantes : il s’agit donc de les comprendre, d’apprendre à les mettre en œuvre et d’estimer leurs performances.
Prérequis
Statistiques, programmation
Contenu
- Mesures de distance, similarité, dissemblance
- Normalisation et transformation
- Classification
- Arbre de décision et forêts aléatoires
- Méthodes bayésiennes
- k plus proches voisins
- Analyse discriminante linéaire
- Clustering
- k means, k medoids
- Clustering hiérarchique
- Méthodes basées sur la densité
- Méthodes basées sur une grille
- Evaluation des performances
- Extraction de règles d'association et d'itemsets fréquents
- Logiciels, librairies et boites à outils disponibles
Références
Data Mining: Concepts and Techniques, J. Han and M. Kamber, 2006
Hadley Wickham & Garret Grolemund, R for Data Science, https://r4ds.had.co.nz
Pedregosa et al., Scikit-learn: Machine Learning in Python, JMLR, 2011 https://scikit-learn.org/
Mots-clés : prétraitement des données ; classification et prédiction ; partitionnement ; caractérisation et discrimination ; règles d’association
Traitement de données post-génomiques
Niveau : Master 1 parcours BBS et BGE
ECTS : 6
Objectifs
Cette unité d'enseignement a pour objectif de fournir une large palette de connaissances et de compétences en bioinformatiques et biostatistiques appliquées à un domaine en constante évolution en biologie moderne: la post-génomique. Cette dernière cherche à étudier la fonction et l'expression des gènes séquencés en génomique par la caractérisation systématique du rôle des mutations, de l'expression des ARN messagers, des ARN non-codants, ainsi que des protéines. Les données générées par la post-génomique sont extrêmement volumineuses et de très grandes dimensions (Big Data). Les applications sont nombreuses: génétique humaine, cancérologie, métagénomique, mais aussi, amélioration des espèces d'intérêt agronomique et étude de l'évolution des espèces.
Prérequis
Connaissances en biologie moléculaire, génomique, biochimie des protéines, ainsi qu'en algorithmique, théorie des graphes et traitement statistique de données.
Contenu
Le premier atelier aborde la manipulation de gros fichiers de séquences, l'alignement des séquences sur le génome, l'assemblage de novo de génomes, la détection de SNP ainsi que leur annotation fonctionnelle. Le second atelier traite de la mesure du niveau d'expression des ARN messagers, de l'analyse différentielle, des méthodes d'enrichissement d'ensemble de gènes et de la détection de variants d'épissage alternatif. Le troisième atelier permet de caractériser les différentes familles d'ARN non codants, d'employer les banques de données et de maîtriser les méthodes bioinformatiques pour traiter les données. Le quatrième atelier présente les caractéristiques générales des spectromètres de masse et les stratégies d'analyse protéomique. Il traite aussi de l'analyse des modifications post-traductionnelles et à celle des interactomes, des stratégies de quantification haut-débit et du traitement des données protéomiques.
Références
Pevsner. Bioinformatics and Functional Genomics, 2015.
Datta and Nettelton. Statistical Analysis of Next Generation Sequencing Data, 2014.
Wu and Choudry, Next Generation Sequencing in Cancer Research, 2013.
Mots Clefs : Haut-débit ; Assemblage de génome ; SNP ; RNA-seq ; small RNA-seq ; Protéomique
Evolution moléculaire
Niveau : Master 1 parcours BBS et BGE
ECTS : 3
Objectifs
Cet enseignement a pour but d'initier les étudiants aux analyses d'évolution moléculaire. Les concepts et méthodes développées dans cette discipline seront détaillés et seront accompagnés par leur mise en pratique sur des cas concrets de reconstruction phylogénétique. L'accent sera mis sur l'interprétation évolutive des arbres obtenus.
Prérequis
Contenu
Cette UE introduira les concepts de l’évolution puis présentera les différents modèles d'évolution des séquences génomiques (ADN et protéines) et les différentes approches de reconstruction d’arbres (parcimonie, méthode de distances, méthode du maximum de vraisemblance). Les méthodes permettant le choix du modèle évolutif le plus adapté aux données traitées, celles utilisées pour analyser la stabilité de la topologie et celles permettant l'analyse de la congruence de plusieurs arbres seront également développées. L'étude de l'impact des forces évolutives (sélection naturelle, dérive,…) sur le polymorphisme des séquences sera aussi abordé. Les concepts et approches vus en cours seront illustrés par des cas concrets (évolution des séquences d'une famille de protéines, pression de sélection sur certains gènes et régions du génome, reconstruction de la phylogénie d'un ensemble d'espèces, etc) lors de séances de TP sur ordinateurs. Au cours des TD, l'accent sera mis sur l'interprétation évolutive des topologies d'arbres (détection des évènements de duplication et/ou perte de gène, de transferts horizontaux etc.)
Références
Concepts et Méthodes en Phylogénie Moléculaire (2010). Guy Perrière et Céline Brochier-Armanet. Collection IRIS, Springer
Computational Molecular Evolution. Ziheng Yang. Oxford University Press.
Mots Clefs : évolution ; méthodes de reconstruction d’arbre phylogénétique ; congruence ; bootstrap ; sélection naturelle ; dérive
Projet tutoré en bioinformatique
Niveau : Master 1 parcours BBS et BGE
ECTS : 3
Objectifs
Mettre en œuvre des techniques de méthodologie de travail et d'organisation de projet.
Prérequis
UE du premier semestre du M1
Contenu
Partie I
Méthodologie de travail en groupe et de suivi de projet. Outils de suivi de modifications et de travail collaboratif. Licences logicielles.
Partie II
Projets en groupes de 3 à 4 personnes. Travail sur des thématiques de bioinformatique mettant en œuvre les concepts et compétences acquises au premier semestre. Utilisation de techniques de travail en groupe et de suivi de projets. Évaluation par des rapports de suivi de projet et une soutenance orale.
Références
PMI, management de projet : un référentiel de connaissances, AFNOR, 2004
Management d'un projet système d'information : principes, techniques, mise en œuvre et outils, Chantal Morley, Dunod, 2006
Mots-clés : méthodologie de travail ; travail en groupe ; suivi de projet ; suivi des modifications ; outils collaboratifs
Introduction Modélisation Moléculaire
Niveau : Master 1 parcours BBS
ECTS : 3
Objectifs
Ce module a pour but d'initier les étudiants aux concepts de base de la modélisation moléculaire. A l'issu de cet enseignement l'étudiant sera capable de créer une molécule et de déterminer sa structure optimale, tant in vacuo que dans un complexe récepteur-ligand.
Prérequis
Contenu
Présentation de concepts de base de la modélisation de structures biomoléculaires. Seront abordés les aspects théoriques et computationnels de la détermination de structures tridimensionnelles de molécules d'intérêt biologique par l'approche empirique, basée sur le champ de force et l'optimisation de géométrie. La partie pratique du module sera consacrée à la création, visualisation, modification et optimisation de structures moléculaires.
Références
Molecular Modelling: Principles and Applications", Andrew Leach, Prentice Hall, 2001.
"Molecular Modelling for Beginners", Alan Hinchliffe, John Wiley & Sons Ltd, 2003.
"Molecular Modeling and Simulation", Tamar Schlick, Springer-Verlag New York Inc., 2010.
Mots Clefs : Visualisation et manipulation de molécules ; structure et dynamique moléculaire ; champs de force ; minimisation d'énergie ; complexe récepteur-ligand
Analyse de données multivariées
Niveau : Master 1 parcours BBS et BGE
ECTS : 3
Objectifs
A l'issue de cet enseignement, les étudiants devront être à même :
- d'organiser des données et de formuler une problématique pertinente
- de choisir la (ou les) méthode(s) d'analyse en fonction de la nature des données et de la problématique formulée
- de mettre en œuvre ces méthodes (utilisation du logiciel R)
- de représenter graphiquement et d'interpréter les résultats
- de rédiger les conclusions dans un rapport de synthèse|
Prérequis
Une UE de biostatistiques élémentaires est exigée, ainsi que des connaissances de base du logiciel R.
Contenu
Cet enseignement propose une présentation des principales méthodes d'analyse adaptées aux données multidimensionnelles. Les méthodes abordées seront illustrées à partir d'exemples réels provenant d'études écologiques. L'enseignement cherchera à montrer plus particulièrement en quoi (1) la nature complexe des systèmes biologiques conduit souvent à la nécessité de prendre en compte un grand nombre de descripteurs, et (2) l'écologie est un champ d'application privilégié des diverses méthodes abordées.
Les aspects théoriques indispensables à la compréhension et à la bonne utilisation de ces méthodes seront traités au cours des CM. Les TP, en salle informatique, permettront aux étudiants de mettre en pratique ces méthodes et donner une place importante à l'interprétation écologique des résultats statistiques. Enfin, un projet réalisé en fin d'UE par binôme permettra aux étudiants d'être confrontés à un problème biologique concret. Ce projet portera sur l'analyse d'un jeu de données collecté dans le cadre d'une étude écologique et aboutira à la rédaction d'un rapport et une présentation orale du travail effectué.
Références
Escoffier & Pagès. Analyses factorielles simples et multiples – Objectifs, méthodes et interprétation. Dunod
Lebart, Piron, & Morineau. Statistiques exploratoire multidimensionnelle. Dunod
Mots Clefs : Analyses multivariées ; structure des tableaux de données en écologie ; liaisons entre descripteurs (biologiques, environnementaux, ...).
Biostatistiques : Utilisation avancée du modèle linéaire
Niveau : Master 1 parcours BBS et BGE
ECTS : 3
Objectifs
A l'issue de cet enseignement, les étudiants devront être à même d'analyser un jeu de données complexe en utilisant des modèles linéaires (lm) ou le des modèles linéaires généralisés (glm). Ils devront maîtriser suffisamment les bases théoriques de ces outils pour pouvoir montrer la pertinence de leur choix d'analyse, et interpréter en détail les résultats obtenus.
Prérequis
Statistiques descriptives, lois de probabilités usuelles, test d'hypothèse, régression linéaire, ANOVA, ANCOVA, utilisation basique du logiciel R.
Contenu
Cet enseignement propose une présentation détaillée des applications du modèle linéaire et du modèle linéaire généralisé à l'analyse des données biologiques. Les éléments théoriques permettant de comprendre les conditions d'application de ces méthodes d'analyse seront expliqués. L'accent sera mis sur les outils permettant de vérifier que ces conditions d'application sont bien remplies, et sur la démarche à adopter lorsqu'elles ne le sont pas.
L'enseignement sera illustré en travaux pratiques par l'analyse détaillée de jeux de données tirés de travaux en écologie, biologie comportementale et biologie évolutive.
Références
The R Book, 2nd Edition. M.J. Crawley 2012. Wiley
Mots Clefs : Statistique ; Modèle linéaire ; Modèle linéaire généralisé
Stage (facultatif)
Niveau : Master 1 parcours BBS et BGE
ECTS : 0
Objectifs
Ce stage optionnel permet aux étudiants d'avoir un premier contact avec le monde professionnel et donc une première immersion dans une équipe de recherche et développement appartenant soit à un laboratoire du milieu académique ou d'une entreprise.
Prérequis
Contenu
Un stage optionnel d'un à deux mois est proposé en fin d'année universitaire car le besoin d'acquérir et/ou de renforcer des compétences dans différents champs disciplinaires (informatique, mathématiques et bioinformatique) ne permet pas d'introduire un stage obligatoire dans le cursus. Il est cependant fortement conseillé d'effectuer ce stage car il permettra d'apprendre à travailler en équipe, d'organiser son travail de manière à répondre aux échéances exigées par l'encadrant, de trouver des solutions pour résoudre des problèmes qui n'avaient pas été envisagés au départ. C'est également l'opportunité de confronter sa formation académique à des problématiques concrètes et de de mettre en oeuvre les compétences acquises. Il constitue une bonne préparation au stage obligatoire de 6 mois de la seconde année de master.
Mots Clefs : Immersion professionnelle
UE Master 2
Bases de données avancées
Niveau : Master 2 parcours BBS
ECTS : 3
Objectifs
Cet enseignement a pour objectif de former les étudiants aux concepts avancés liés aux bases de données relationnelles : évaluation d'une requête relationnelle, interrogation d'une base de données via un langage procédural, administration d'une base de données.
Prérequis
Les concepts fondamentaux en bases de données (modèles entité/association, modèle relationnel, notions fondamentales en SQL)
Contenu
D'abord, les différentes étapes pour l'évaluation d'une requête relationnelle dans un environnement centralisé sont passées en revue. Ensuite, une introduction à un langage procédural est faite afin d'interroger et d’administrer une base de données.
Références
Georges Gardarin - Bases de Données - EYROLLES
Mots Clefs : Bases de données ; Système de gestion de bases de données ; PL/SQL; Vues relationnelles.
Introduction Apprentissage automatique
Niveau : Master 2 parcours BBS et BGE
ECTS : 3
Objectifs
L'apprentissage automatique ou apprentissage artificiel est un champ d'étude de l'intelligence artificielle qui se fonde sur des approches mathématiques et statistiques pour donner aux ordinateurs la capacité d'« apprendre » à partir de données, c'est-à-dire d'améliorer leurs performances à résoudre des tâches sans être explicitement programmés pour chacune. Plus largement, il concerne la conception, l'analyse, l'optimisation, le développement et l'implémentation de telles méthodes. Les progrès récents de ce champ de l'IA font qu'il s'introduit dans tous les champs scientifiques, et en particulier la biologie et la génétique.
Le but du module est donc de donner aux étudiant(e)s un aperçu global de l'apprentissage, à la fois l'apprentissage par renforcement et l'apprentissage à partir d'exemples. Pour ces 2 sous domaines, les principes théoriques sont introduits, des algorithmes sont présentés. La partie pratique consiste en l'implémentation en python de ces algorithmes ainsi que la manipulation des librairies essentielles au domaine (Keras, tensorflow, scikit-learn).
A la fin du module, les étudiant(e)s sont capables d'analyser un jeu de données, de le préparer, de choisir un algorithme d'apprentissage adapté et de l'implémenter.
Prérequis
Calcul différentiel; Probabilités (les fondamentaux seulement)
Contenu
Apprentissage par renforcement
- Définition
- Equation de Bellman
- Q-table – implémentation en python
Apprentissage à partir d’exemples
- Principes génériques et fonction de perte
- Minimisation et descente en gradient
- Réseau de neurones
- Re-implémentation de la Q-table via réseau de neurones
- Convolution pour traitement d’images
- Problèmes et solutions : vanishing gradient, overfitting, etc.
- SVM
Références
Machine learning (1997) Tom M. Mitchel, Publisher: McGraw-Hill
Mots Clefs : Apprentissage automatique ; Règles d’association ; Réseau de neurones multi-couches
Gestion de données non structurées et applications post-génomiques
Niveau : Master 2 parcours BBS
ECTS : 3
Objectifs
Ce module a pour but d'enseigner aux étudiant·e·s comment modéliser, représenter et exploiter les masses de données complexes et variées disponibles dans les données publiques et/ou issues des approches de post-génomiques.
Prérequis
mathématiques ; statistiques ; traitement de graphes ; fouille de données ; bases de données relationnelles
Contenu
Introduction aux systèmes de gestion de données non structurées (NoSQL).
Modélisation, stockage et interrogation de données selon plusieurs modèles NoSQL (orienté documents et orienté graphe).
Alimentation de la base de données par des sources de données hétérogènes.
Exploitation de cette base par des exemples de projets de post-génomique comme par exemple la caractérisation d'un ensemble de gènes ou la priorisation de gènes candidats.
Références
Christian Theil Have, Lars Juhl Jensen, Are graph databases ready for bioinformatics?, Bioinformatics, Volume 29, Issue 24, 15 December 2013, Pages 3107–3108 https://doi.org/10.1093/bioinformatics/btt549
Polyane Wercelens et al., Bioinformatics Workflows With NoSQL Database in Cloud Computing, Evol Bioinform Online, 2019
Les bases de données NoSQL et le BigData : Comprendre et mettre en oeuvre Ed. 2. Auteur: Bruchez, Rudi, Eyrolles (2015). ISBN: 978-2-212-14155-9
Bases de données orientées graphes avec Neo4j - Manipuler et exploiter vos bases de données orientées graphes. Auteurs: Benhenni, Amine Lies,Bois, François-Xavier. Eyrolles (2016). ISBN: 978-2-212-13804-7
Mots-clés : masses de données non structurées ; données hétérogènes ; caractérisation d'un ensemble
Phylogénomique
Niveau : Master 2 parcours BBS et BGE
ECTS : 3
Objectifs
La phylogénomique est une nouvelle discipline à l’intersection de l’évolution moléculaire et de la génomique. Elle permet de reconstruire l’histoire évolutive des espèces au travers de leurs génomes et ainsi de fournir un cadre conceptuel dans lequel l’évolution des gènes et des organismes peut être étudiée. L’identification des gènes orthologues entre espèces contribue à la prédiction de la fonction des gènes par transferts d’annotation. L’analyse des répertoires de gènes d’organismes se développant dans des environnements différents permet d’identifier les gènes impliqués dans l’adaptation à des niches écologiques. Cette approche a une contribution majeure pour comprendre la mise en place de résistances aux antibiotiques et l’adaptation des bactéries à leurs hôtes. Elle est en plein essor avec la prolifération des données issues de l’environnement et du milieu médical et grâce aux progrès réalisés pour analyser des gros volumes de données par des méthodes bioinformatiques. Le but de cet enseignement est d’initier les étudiant(e)s aux approches phylogénomiques en détaillant les principes théoriques des méthodes impliquées dans les différentes étapes de l’analyse. La partie pratique consistera à développer une chaîne de traitements en s’appuyant sur une question biologique particulière utilisant des données « réelles » issues de la littérature. A l’issue de cette UE, les étudiant(e)s sont capables de conduire un même type d’analyse sur des données différentes.
Prérequis
Les UE d’Evolution Moléculaire, de Bioinformatique pour la Génomique et de Traitement de graphes et réseaux biologiques.
Contenu
Les différentes approches, de la théorie à la pratique, seront introduites au travers de cours. La mise en œuvre se fera dans le cadre de séances de travaux pratiques utilisant un cluster de calcul.
Les points suivant seront abordés :
- Construction du jeu de données en fonction des objectifs avec une sélection des génomes sur la base de leur qualité (complétude et contamination) et de leur représentativité (réduction de la redondance et optimisation de la diversité).
- Evolution de la structure des génomes (duplication, délétions, inversions, transposition…) par alignements de génomes et par des méthodes basées sur des graphes.
- Evolution du répertoire des gènes (duplication, délétion, recombinaison, conversion, transfert de gènes) et identification des liens d’homologies, d’orthologies et de paralogies.
- Construction de groupes de gènes orthologues entre deux et plus de deux génomes.
- Identification du génome cœur, génome accessoire, et pangénome pour une espèces représentée par plusieurs souches.
- Construction d’un arbre espèce par différentes approches.
- Utilisation de cet arbre pour réconcilier les arbres gènes pour identifier les duplications, pertes et transferts de gènes.
- Utilisation de cet arbre pour la reconstruction d’états ancestraux pour des données discrètes et continues.
Les TP permettront de mettre en pratique les approches et démarches théoriques décrites en cours sur des données « réelles » issues de la littérature. Au cours de ces TP, les étudiants construiront une chaîne de traitements pour automatiser les différentes étapes d’une analyse phylogénomique. Comme exemples de modèles d’études, nous pouvons citer (i) l’étude de l’évolution des génomes des espèces de Prochlorococcus (un genre de cyanobactéries marines photosynthétiques) en lien avec l’adaptation des différents clades de Prochlorococcus à différentes niches écologiques et (ii) l’étude de l’évolution des génomes des différentes souches de Staphylococcus aureus en lien avec leur capacité à franchir la barrière d’espèce et à acquérir des résistances aux antibiotiques.
Références
Mots Clefs: phylogénomique ; évolution ; orthologie ; génomique comparative
Métagénomique écologique et évolutive
Niveau : Master 2 parcours BGE
ECTS : 9
Objectifs
S’approprier les approches :
- de génomique des populations qui permettent d’évaluer l’influence des pressions évolutives (sélection naturelle, dérive génétique, recombinaison, mutation, et migration) sur la diversité génétique d’une espèce.
- de génomique écologique, qui permettent d’identifier les bases génétiques (i) des caractères adaptatifs et (ii) de la réponse des populations à la variabilité de l’environnement biotique et abiotique.
- de métagénomique qui permettent de décrire la diversité taxonomique et fonctionnelle, de modéliser les interactions entre les différentes composantes, et d’émettre des hypothèses sur leur évolution et leur capacité d’adaptation dans un contexte d’environnement changeant.
- de paléogénomique qui permettent de comprendre l’évolution démographique et adaptative de certains groupes taxonomiques sous un angle temporel.
Exploiter des données génomiques de polymorphismes moléculaires provenant d’échantillons populationnels ou environnementaux afin de tester des hypothèses dans le cadre de ces approches, par l’utilisation de logiciels de modélisation (méthodes statistiques descriptives et inférentielles).
Interpréter les résultats issus de l’analyse de données génomiques populationnelles ou environnementales - qu’ils soient originaires d’un article ou des analyses menées par les étudiants.
Prérequis
Les UE de M1 BGE : Génomique Environnementale, Ecologie Evolutive
Contenu
Les différentes approches, de la théorie à la pratique, seront introduites au travers de CM, séminaires et TD de chercheurs et d’enseignants-chercheurs, en s’appuyant sur des projets de recherche existants. Les TD pourront s’appuyer sur l’analyse d’articles méthodologiques ou de recherche ciblant certaines thématiques. La mise en application sur les 4 grandes thématiques se fera dans le cadre de séances de travaux pratiques et de mini-projets, par l’analyse et l’interprétation des données :
- Génomique des populations : détection de locus sous sélection, structuration génétique, inférences de paramètres démographiques.
- Génomique écologique : analyses d’association génotype-phénotype (ex : résistance à un pathogène) et génotype-environnement.
- Métagénomique (thématiques ciblées): détection de pathogènes dans des échantillons (alimentaires, archéologiques), quantification de la biodiversité dans des environnements cibles (systèmes aquatiques, sols,…), métagénomique des interactions (communautés, pollinisateurs).
- Paléogénomique : caractérisation des polymorphismes d’ADN anciens, estimation des phénomènes démographiques anciens (introgression, hybridation, …), détection de locus impliqués dans l’évolution adaptative d’un taxon.
Références
Mots Clefs : génomique des populations, génomique écologique, métagénomique, paléogénomique, Next-Generation Sequencing, Single Nucleotide Polymorphism (SNP), modélisation
Biologie des systèmes 1
Niveau : Master 2 parcours BBS
ECTS : 6
Objectifs
Cet enseignement a pour but d'initier les étudiants à la biologie des systèmes dont l'objectif est de caractériser les composants élémentaires d'un système biologique (par exemple, une voie de régulation, une cellule, un écosystème etc.) pour mettre à jour les propriétés qui résultent de leurs interactions, ceci afin de mieux comprendre le comportement dynamique du système dans sa globalité. Dans cette UE, les problèmes de biologie des systèmes seront abordés via l’approche descendante ou top down (versus l’approche bottom up), qui s'appuie largement sur les résultats expérimentaux obtenus par les approches à haut débit. La compréhension d’un système va donc nécessiter d’intégrer les données obtenues à partir de différentes approches Omics, en fonction de la question abordée. Les concepts et méthodes nécessaires à l’analyse de ces différents types de données Omics seront détaillés et seront accompagnés par leur mise en pratique sur des cas concrets.
Prérequis
UE Traitement des données post-génomiques.
Contenu
Différentes problématiques scientifiques d’actualité en relation avec la biologie des systèmes seront abordées sous forme d'ateliers ce qui requerra une participation active des étudiants comme par exemple la préparation de l'atelier par la lecture et la présentation orale de publications scientifiques sur le sujet, la réalisation de projets personnels ou en groupe, etc.
Parmi les problématiques qui seront abordées, nous pouvons citer l’épigénomique, la métabolomique, l’étude des interactions ADN/protéines à l’échelle du génome, l’organisation spatiale de la chromatine dans la cellule, les approches « single cell ». Cette liste n’est pas exhaustive et pourra évoluer d’année en année en fonction de l’évolution de la discipline. Les concepts et méthodes nécessaires au traitement de ces nouveaux types de données Omics seront détaillés et seront accompagnés par leur mise en pratique pour aborder, par exemple, des questions en recherche biomédicale.
Références
Mots Clefs : Biologie des systèmes ; intégration des données ; traitement des données ChIP-Seq ; Chromosome conformation capture ; épigénomique ; métabolomique ; single cell.
Biologie des systèmes 2
Niveau : Master 2 parcours BBS
ECTS : 6
Objectifs
Dans cette UE, les problèmes de biologie des systèmes seront abordés via l’approche ascendante ou bottom up (versus l’approche top down) dont le but est d’intégrer dans un modèle mathématique l’ensemble des données expérimentales obtenues sur un système d’étude pour pouvoir simuler le comportement dynamique de ce système au cours du temps mais aussi dans différentes conditions physiologiques et/ou environnementales. Les différents formalismes mathématiques disponibles pour modéliser un système biologique et le simuler seront décrits du point de vue théorique et mis en pratique sur des cas concrets de modélisation de processus biologiques. A l’issue de cette UE, les étudiant(e)s sont capables de construire un modèle, de choisir le bon formalisme mathématique en fonction des données expérimentales disponibles sur le processus (système) étudié et de simuler son comportement.
Prérequis
Notions de calcul matriciel; équations différentielles ordinaires
Contenu
Les enseignements aborderont la description des différentes méthodes d'analyse dynamique des réseaux biologiques et de prédiction de leur comportement. Les approches qualitatives ainsi que les approches quantitatives seront abordées. Dans le cas des approches qualitatives, la modélisation par équations différentielles linéaires par morceaux et les modèles logiques seront plus particulièrement développés. Pour les approches quantitatives, les modèles déterministes basés sur les équations différentielles ordinaires seront privilégiés. Les réseaux de Pétri, proposant un formalisme graphique et mathématique permettant de modéliser et simuler le comportement dynamique d'un système par une approche qualitative ou quantitative seront également décrits.
Le problème d’estimation des valeurs des paramètres du modèle quand elles ne sont pas connues expérimentalement sera abordé et quelques méthodes d’estimation de ces paramètres seront décrites.
Les TP aborderont des cas pratiques issus de divers domaines de la biologie comme : les réactions enzymatiques, la croissance de biomasse, l'analyse de processus périodiques et la modélisation de réseaux de régulation de gènes.
Références
An introduction to Systems Biology: Design principles of Biological Circuits (2007). Uri Alon. Chapman & Hall/CRC
Mots Clefs : Biologie des systèmes ; systèmes biologiques dynamiques ; réseaux d'interactions ; modélisation ; simulation
Ouverture : international, bioéthique, insertion professionnelle
Niveau : Master 2 parcours BBS et BGE
ECTS : 3
Objectifs
Les objectifs de cette UE sont, d’une part, ouvrir l’esprit des futurs diplômés à des notions qui ne sont pas leur cœur disciplinaire et, d’autre part, développer les compétences transversales nécessaires à une insertion professionnelle réussie.
Prérequis
Contenu
Les notions suivantes seront abordées à la fois sous forme d'enseignements traditionnels et de séminaires, y compris par des conférenciers internationaux :
- la bioéthique (en particulier nos droits et devoirs envers les organismes vivants, y compris les plus simples, le rapport du scientifique au public);
- intelligence artificielle : pouvoir et limites (notamment en matière d'éthique);
- les avancées les plus récentes dans le domaine des nanobiotechnologies : bases, concepts, applications.
Enfin, en vue de leur insertion professionnelle, des tables rondes seront organisées avec d'anciens diplômés du Master, recrutés dans le monde académique ou dans le secteur privé : recherche d'emploi, stratégies de candidature, intégration, mise en réseau. Les étudiants seront interpellés à la fois par un questionnement personnel sur leurs objectifs professionnels et par des entretiens d'embauche fictifs.
Références
Mots Clefs :
Communication scientifique
Niveau : Master 2 parcours BBS et BGE
ECTS : 3
Objectifs
L’objectif est d'apprendre et de mettre en œuvre la synthèse et la présentation de travaux scientifiques via différents supports de communication.
Prérequis
Contenu
Elaboration de support de communication : diapositives, présentations interactives, poster.
Rédaction d'une synthèse de l'état de l'art sous forme d'une revue en anglais respectant un format de journal international.
En pratique, chaque étudiant se verra proposer une publication scientifique en relation avec les thématiques abordées dans les UE de biologie des systèmes 1 et 2 (parcours BBS), de méta-génomique écologique et évolutive (parcours BGE)et de phylogénomique. L'étudiant devra présenter cette publication à l'oral avec un support écrit en anglais. Le thème de cette publication servira de point de départ pour la synthèse et la rédaction de la mini-revue dont le format suivra celui demandé pour l'écriture d'une revue dans un journal international.
Références
Mots Clefs : analyse critique de publication ; rédaction scientifique ; recherche bibliographique ; présentation orale
Stage académique
Niveau : Master 2 parcours BBS et BGE
ECTS : 30
Objectifs
Le stage de 6 mois (de janvier à juin) en milieu académique permettra à l'étudiant d'apprendre à travailler en équipe, de gérer un projet de recherche et donc d'organiser son travail de manière à répondre aux échéances exigées par l'encadrant, de trouver des solutions pour résoudre des problèmes qui n'avaient pas été envisagés au départ. Il permettra aussi de mettre en oeuvre les compétences acquises lors de la formation académique pour résoudre des problématiques concrètes.
Le travail réalisé donnera lieu à un rapport écrit et à une soutenance orale.
Prérequis
Contenu
Travailler dans un environnement pluridisciplinaire (biologistes, bioinformaticiens, biostatisticiens, informaticiens) au sein d'une entreprise tout en faisant preuve d'autonomie et d'initiatives.
Défendre un projet et/ou des résultats en utilisant les moyens de communication adéquats lors de réunions d'équipe, conférences ou jurys.
Mots Clefs : immersion professionnelle.
Stage professionnel
Niveau : Master 2 parcours BBS et BGE
ECTS : 30
Objectifs
Le stage professionnel de 6 mois (de janvier à juin) en entreprise ou milieu académique permettra à l'étudiant d'apprendre à travailler en équipe, de gérer un projet de développement logiciel lié à une question biologique et donc d'organiser son travail de manière à répondre aux échéances exigées par l'encadrant, de trouver des solutions pour résoudre des problèmes qui n'avaient pas été envisagés au départ. Il permettra aussi de mettre en oeuvre les compétences acquises lors de la formation académique pour résoudre des problématiques concrètes.
Le travail réalisé donnera lieu à un rapport écrit et à une soutenance orale.
Prérequis
Contenu
Travailler dans un environnement pluridisciplinaire (biologistes, bioinformaticiens, biostatisticiens, informaticiens) au sein d'une entreprise tout en faisant preuve d'autonomie et d'initiatives.
Défendre un projet et/ou des résultats en utilisant les moyens de communication adéquats lors de réunions d'équipe, conférences ou jurys.
.
Mots Clefs : immersion professionnelle.
Historique de la formation
- 1999 : Création du DESS de Bioinformatique
- 2005 : Réforme LMD, le DESS devient un Master 2 Professionnel
- 2011 : Evolution du Master sur deux années de formation M1-M2I au sein de la mention Microbiologie-AgroBiosciences-Biologie des Systèmes (MABS)
- 2016 : Mention Bioinformatique parcours Bioinformatique et Biologie des Systèmes
- 2022 : Création d'un second parcours Bioinformatique et Génétique Environnementale au sein de la mention Bioinformatique
Jusqu’en 2015 la mention Bioinformatique était enregistrée sous la dénomination mention Microbiologie-AgroBiosciences-Biologie des Systèmes (MABS) et était constitué par le parcours Bioinformatique et Biologie des Systèmes en première année de master et par la spécialité Bioinformatique et Biologie des Systèmes en deuxième année de master.